Армирование фундаментов снип: СП 63.13330.2018 Бетонные и железобетонные конструкции. Основные положения. СНиП 52-01-2003 (с Изменением N 1)
- Армирование ленточного фундамента
- Армирование ленточного фундамента СНИП — bmi-rus.ru
- Схема армирования ленточного фундамента по СНиП
- Для чего нужно армировать фундамент
- Требования СНиП к монтажу арматуры
- Как правильно армировать ленточный фундамент
- Как армировать фундамент столбчатой конструкции
- Армирование плитной конструкции фундамента
- Снип фундаменты ленточные
- Схема армирования и технология строительства основания
- Заливка бетона в траншею
- Калькулятор вес арматуру
- Как проверяется прочность бетона
- Создание ленточного фундамента по нормам СНИП
- Определение по Снип
- Нормативно-законодательная база
- Нормы СНИП к арматуре
- Этапы строительства ленточного фундамента по СНИП
- Армирование ленточного фундамента – правила, схемы, инструкции
- Расчет необходимого количества материалов
- Особенности армирования ленточного фундамента
- Для чего нужно армировать ленточный фундамент?
- Особенности конструкции армирующего каркаса
- СП 15. 13330.2012 «Каменные и армокаменные конструкции. Актуализированная редакция СНиП II-22-81»
- Frontiers | Генотипирование путем секвенирования Идентификация SNP для сельскохозяйственных культур без эталонного генома: использование картирования на основе транскриптомов в качестве альтернативной стратегии
- SNP (однонуклеотидный полиморфизм) | DNAeXplained — Генетическая генеалогия
- 027 mtDNAb:
8 HV9
Образец: VK57 / Gotland_Frojel-03601 Местоположение: Frojel, Gotland, Швеция Возраст: Viking 900-1050 CE Y-ДНК: R-L151 mtDNA: J1c6
Образец: VK60 / Gotland_Frojel-00702 Местоположение: Frojel, Gotland, Швеция Возраст: Viking 900-1050 CE Y-ДНК: R-YP1026 mtDNA: h23a1a1b
8
Образец: VK64 / Gotland_Frojel-03504 Местоположение: Frojel, Gotland, Швеция Возраст: Viking 900-1050 CE Y-ДНК: R-BY58559 mtDNA: I1a1
Образец: VK70 / Дания_Tollemosegard-EW Местоположение: Толлемосегард, Силенд, Дания Возраст: Ранний викинг Поздний германский железный век / ранний викинг Y-ДНК: I-BY73576 mtDNA4:
Образец: VK71 / Дания_Tollemosegard-BU Местоположение: Толлемосегард, Силенд, Дания Возраст: Ранний викинг Поздний германский железный век / ранний викинг Y-ДНК: I-S22349 mtDNA1:
Образец: VK75 / Гренландия, поздний 0929 Местоположение: V051, Западное поселение, Гренландия Возраст: Поздний скандинавский 1300 г. н.э. Y-ДНК: R-P310 мтДНК: H54
Образец: VK87 / Дания_Hesselbjerg Grav 41b, sk PC Местоположение: Hesselbjerg, Ютландия, Дания Возраст: Viking 850-900 CE Y-ДНК: R-Z198
027 mtDNA2: K1
Образец: VK95 / Iceland_127 Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 века н.э. Y-ДНК: R-S658 mtDNA: H6a1a3a
Образец: VK98 / Iceland_083 Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 века н.э. Y-ДНК: I-BY3433 FTDNA Комментарий: Разделяет I-BY3430.Получено для 1 предка для 6. Новый путь = I-BY3433> I-BY3430 мтДНК: T2b3b
Образец: VK101 / Iceland_125 Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 вв. Н. Э. Y-ДНК: R-BY110718 мтДНК: U5b1g
Образец: VK102 / Iceland_128 Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 веков н.э. Y-ДНК: R-Y96503 FTDNA Комментарий: Делится 3 SNP с мужчиной из Швеция.Образует новую ветвь после R-FGC23826. Новая ветвь = R-Y96503 мтДНК: J1c3f
Образец: VK110 / Iceland_115S Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 вв. Н. Э. Y-ДНК: I-FGC21682 мтДНК: h20-x
Образец: VK117 / Norway_Trondheim_SK328 Местоположение: Тронхейм, Нор-Мид, Норвегия Возраст: Средневековье 12-13 века н.э. Y-ДНК: R-S9257 мтДНК:
0 h2a3a 9
Образец: VK123 / Iceland_X104 Местоположение: Хофстадир, Исландия Возраст: Викинг 10-13 вв. Н. Э. Y-ДНК: R-Y130994 FTDNA Комментарий: Делится 17 SNP с мужчиной из ОАЭ.Создает новую ветвь после R2-V1180. Новая ветвь = R-Y130994 мтДНК: J1c9
Образец: VK127 / Iceland_HDR08 Местоположение: Хрингсдалур, Исландия Возраст: Викинг 10 век н.э. Y-ДНК: R-BY мтДНК: h4g1b
Образец: VK129 / Iceland_ING08 Расположение: Ингиридарстадир, Исландия Возраст: Викинг 10 век н.э. Y-ДНК: R-BY154143 Комментарий FTDNA: Делится 3 SNP с мужчиной из Швеции.Образует новую ветвь после R1a-YP275. Новая ветвь = R-BY154143 мтДНК: U5b1b1a
Образец: VK133 / Дания_Galgedil KO Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 8-11 века н.э. Y-ДНК: R-Z8 мтДНК: K1a4a1a3
Образец: VK134 / Дания_Galgedil ALZ Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: R-BY97519 мтДНК: h2cg
Образец: VK138 / Дания_Galgedil AQQ Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 9-11 век н.э. Y-ДНК: R-S1491 мтДНК: T2b5
Образец: VK139 / Дания_Galgedil ANG Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-BY32008 мтДНК: J1c3k
Образец: VK140 / Дания_Галгедил PT Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: G-M201 мтДНК: h37f
Образец: VK143 / UK_Oxford_ # 7 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-Y13833 FTDNA Комментарий: Разделяет R-Y13816.Получено для 6 предков для 3. Новый путь = R-Y13816> R-Y13833 мтДНК: U5b1b1-T16192C!
Образец: VK144 / UK_Oxford_ # 8 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-Y2592 mtDNA1:
Образец: VK145 / UK_Oxford_ # 9 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-YP1708 mtDNA:
Образец: VK146 / UK_Oxford_ # 10 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-M6155 mtDNA3:
Образец: VK147 / UK_Oxford_ # 11 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-Y75899 mtDNA1q
Образец: VK148 / UK_Oxford_ # 12 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-M253 mtDNA1a:
Образец: VK149 / UK_Oxford_ # 13 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-M253 htDNA1:
Образец: VK150 / UK_Oxford_ # 14 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-FT4725 htDNA: mtDNA: -C16239T
Образец: VK151 / UK_Oxford_ # 15 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-S19291 mtDNA4: -T152C!
Образец: VK153 / Poland_Bodzia B1 Местоположение: Бодзя, Польша Возраст: Викинг 10-11 века н.э. Y-ДНК: R-M198 мтДНК: h2c3
Образец: VK156 / Poland_Bodzia B4 Расположение: Бодзия, Польша Возраст: Викинг 10-11 веков н.э. Y-ДНК: R-Y9081 мтДНК: J1c2c2a
Образец: VK157 / Poland_Bodzia B5 Местоположение: Бодзия, Польша Возраст: Викинг 10-11 веков н.э. Y-ДНК: I-S2077 мтДНК: h2c
Образец: VK159 / Russia_Pskov_7283-20 Местоположение: Псков, Россия Возраст: Викинг 10-11 веков н.э. Y-ДНК: R-A7982 mtDNA: U2e2a1d
Образец: VK160 / Russia_Kurevanikka_7283-3 Местоположение: Куреваниха, Россия Возраст: Викинг 10-13 века н.э. Y-ДНК: R-YP1137 мтДНК: C4a1a-T195C
Образец: VK163 / UK_Oxford_ # 1 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-M253 mtDNA2 :a
Образец: VK165 / UK_Oxford_ # 3 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-S18218 mtDNA1:
Образец: VK166 / UK_Oxford_ # 4 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-BY67003 FTDNA Комментарий: Сплит R-BY45170 (DF27).Получено для 2, предков для 7. Новый путь = R-BY67003> R-BY45170 мтДНК: h4ag
Образец: VK167 / UK_Oxford_ # 5 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-BY34674 8 mtDNA1
Образец: VK168 / UK_Oxford_ # 6 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-Z18 mtDNA1a mtDNA1a
Образец: VK170 / Isle-of-Man_Balladoole Местоположение: Balladoole, IsleOfMan Возраст: Викинг 9-10 веков н.э. Y-ДНК: R-S3201 mtDNA:
05 HV9b 9b 9b
Образец: VK172 / UK_Oxford_ # 16 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-FT7019 mtDNA1:
Образец: VK173 / UK_Oxford_ # 17 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-FT13004 FTDNA Комментарий: Расщепляет I2-FT12648, полученный для 5, наследственный для 7.Новый путь FT13004> FT12648 мтДНК: U5a1b-T16362C
Образец: VK174 / UK_Oxford_ # 18 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-FGC17429 mtDNA -C16239T
Образец: VK175 / UK_Oxford_ # 19 Местоположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-Y47841 FTDNA Комментарий: Делит 6 SNP с человеком из Швеции, ниже R-BY38950 (R-Y47841) мтДНК: h2a1
Образец: VK176 / UK_Oxford_ # 20 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: I-FT3562 mtDNA: mtDNA:
Образец: VK177 / UK_Oxford_ # 21 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-FT31867 FTDNA Комментарий: Делится 3 SNP с мужчиной из Греции.Образует новую ветвь после R-BY220332 (U152). Новая ветвь = R-FT31867 мтДНК: H82
Образец: VK178 / UK_Oxford_ # 22 Расположение: St_John’s_College_Oxford, Oxford, England, UK Возраст: Viking 880-1000 CE Y-ДНК: R-BY176639 FTDNA Комментарий: Соединяется с PGA3 (Проект персонального генома, Австрия) и клиентом FTDNA из Дании. Клиенты PGA и FTDNA сформировали филиал ранее на этой неделе, VK178 присоединится к ним по адресу R-BY176639 (под L48) мтДНК: K2a5
Образец: VK179 / Гренландия F2 Расположение: Ø029a, Восточное поселение, Гренландия Возраст: Раннее скандинавское население 10–12 веков н. Э. Y-ДНК: I-F3312 мтДНК: K1a3a
Образец: VK183 / Гренландия F6 Расположение: Ø029a, Восточное поселение, Гренландия Возраст: Ранние скандинавские 10-12 вв. Н.э. Y-ДНК: I-F3312 мтДНК: T2b21
Образец: VK184 / Гренландия F7 Расположение: Ø029a, Восточное поселение, Гренландия Возраст: Ранние скандинавские 10-12 вв. Н. Э. Y-ДНК: R-YP4342 мтДНК: h5a1a4b
Образец: VK186 / Гренландия KNK- [6] Местоположение: Ø64, Восточное поселение, Гренландия Возраст: Ранние скандинавские 10-12 века н.э. Y-ДНК: I-Y79817 FTDNA Комментарий : Делится 3 SNP с мужчиной из Норвегии ниже I-Y24625.Новая ветвь = I-Y79817 мтДНК: h2ao
Образец: VK190 / Гренландия, поздний-0996 Местоположение: Ø149, Восточное поселение, Гренландия Возраст: Поздний скандинавский 1360 г. н.э. Y-ДНК: I-FGC15543 FTDNA Комментарий: Сплит I- FGC15561. Получено 11 предков для 6. Новый путь = I-FGC15543> I-FGC15561 мтДНК: K1a-T195C!
Образец: VK201 / Orkney_Buckquoy, sk M12 Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK Возраст: Викинг 5-6 века н.э. Y-ДНК: I-B293 mtDNA1
Образец: VK202 / Orkney_Buckquoy, sk 7B Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK Возраст: Viking 10 век н.э. Y-ДНК: R-A151 mtDNA1
Образец: VK203 / Orkney_BY78, Ar.1, sk 3 Расположение: Brough_Road_Birsay, Оркни, Шотландия, Великобритания Возраст: Викинг 10 век н.э. Y-ДНК: R-BY10450 FTDNA Комментарий: FT83323-
0 mtDNA1 9a a h5a
Образец: VK204 / Orkney_Newark for Brothwell Местоположение: Newark_Deerness, Orkney, Scotland, UK Возраст: Викинг 10 век н.э. Y-ДНК: R-BY115469 mtDNA 9:
8 h2
Образец: VK205 / Orkney_Newark 68/12 Расположение: Newark_Deerness, Orkney, Scotland, UK Возраст: Викинг 10 век н.э. Y-ДНК: R-YP4345 mtDNA:
8 h4
Образец: VK210 / Poland_Kraków-Zakrzówek gr.24 Местоположение: Краков, Польша Возраст: Средневековье 11-13 вв. Н. Э. Y-ДНК: I-Z16971 мтДНК: H5e1a1
Образец: VK211 / Польша_Cedynia gr. 435 Местоположение: Cedynia, Poland Возраст: Средневековье 11-13 вв. Н. Э. Y-ДНК: R-M269 мтДНК: W6
Образец: VK212 / Польша_Cedynia gr. 558 Расположение: Сединия, Польша Возраст: Викинг 11-12 века н.э. Y-ДНК: R-CTS11962 мтДНК: h2-T152C!
Образец: VK215 / Дания_Гердруп-Б; sk 1 Местоположение: Гердруп, Силенд, Дания Возраст: Викинг 9 век н.э. Y-ДНК: R-M269 мтДНК: J1c2k
Образец: VK217 / Sweden_Ljungbacka Местоположение: Ljungbacka, Мальме, Швеция Возраст: Викинг 9-12 вв. Н.э. Y-ДНК: R-L151 мтДНК: J1b1b1
Образец: VK218 / Russia_Ladoga_5680-4 Расположение: Ладога, Россия Возраст: Викинг 10-12 вв. Н.э. Y-ДНК: R-BY2848 мтДНК: H5
Образец: VK219 / Russia_Ladoga_5680-10 Местоположение: Ладога, Россия Возраст: Викинг 10-11 века н.э. Y-ДНК: I-Y22024 mtDNA: T2b6a
Образец: VK220 / Russia_Ladoga_5680-11 Местоположение: Ладога, Россия Возраст: Викинг 10-12 вв. Н.э. Y-ДНК: I-FT253975 FTDNA Комментарий: CTS2208 +, BY47171-, CTS7676-, Y20288-, BY69785-, FT253975 + мтДНК: J2b1a
Образец: VK221 / Russia_Ladoga_5757-14 Местоположение: Ладога, Россия Возраст: Викинг 9-10 вв. Н.э. Y-ДНК: I-Y5473 мтДНК: K1d
Образец: VK223 / Россия_Гнездово 75-140 Местоположение: Гнездово, Россия Возраст: Викинг 10-11 вв. Н.э. Y-ДНК: I-BY67763 мтДНК: h23a1a1c
Образец: VK224 / Россия_Гнездово 78-249 Местоположение: Гнездово, Россия Возраст: Викинг 10-11 веков н.э. Y-ДНК: N-CTS2929 мтДНК: H7a1
Образец: VK225 / Iceland_A108 Расположение: Хофстадир, Исландия Возраст: Викинг 10-13 вв. Н. Э. Y-ДНК: R-BY мтДНК: h4v-T16093C
Образец: ВК232 / Готланд_Коппарсвик-240.65 Расположение: Коппарсвик, Готланд, Швеция Возраст: Викинг 900-1050 CE Y-ДНК: R-Y16505 FTDNA Комментарий: Спекулятивное размещение — U106 +, но U106 (C> T) в древние образцы могут ввести в заблуждение. LAV010, NA34, I7779, ble007, R55 и EDM124 — все это древние образцы, не относящиеся к R и являющиеся U106 +. Более консервативное размещение — R-P310 мтДНК: N1a1a1
Образец: VK234 / Faroe_2 Расположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-FT381000 FTDNA Комментарий: То же разделение, что и VK25.У них общий маркер FT381000 (26352237 T> G) мтДНК: h4a1a
Образец: VK237 / Faroe_15 Местоположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-S6355 mtDNA: J2a2c
Образец: VK238 / Faroe_4 Местоположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-YP396 mtDNA: h4a1a
Образец: VK239 / Faroe_5 Местоположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-M269 mtDNA: H5
Образец: VK242 / Faroe_3 Местоположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-S764 mtDNA: h4a1a
Образец: VK244 / Faroe_12 Расположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-CTS4179 mtDNA: h3a2a2
Образец: VK245 / Faroe_16 Расположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: R-BY202785 FTDNA Комментарий: Образует ветвь с VK46 вниз из R-BY202785 (Z287).Новая ветвь = R-FT383000 мтДНК: h4a1
Образец: VK248 / Faroe_22 Местоположение: Church3, Фарерские острова Возраст: Раннее Новое время 16-17 веков н.э. Y-ДНК: I-M253 мтДНК: h59a
Образец: VK251 / Gotland_Kopparsvik-30.64 Местоположение: Коппарсвик, Готланд, Швеция Возраст: Viking 900-1050 CE Y-ДНК: R-M459 mtDNA: 9 U5b1e
Образец: VK256 / UK_Dorset-3722 Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK Возраст: Viking 10-11 веков н.э. Y-ДНК: R-YP5718 m
Образец: VK257 / UK_Dorset-3723 Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э. Y-ДНК: I-Y19934 ma
Образец: VK258 / UK_Dorset-3733 Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э. Y-ДНК: R-YP1395 FTDNA Комментарий: FTDNA 5 SNP с мужчиной из Норвегии.Образует новую ветвь R-YP1395. Новая ветвь = R-Ph520 мтДНК: K1a4a1
Образец: VK259 / UK_Dorset-3734 Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK Возраст: Viking 10-11 веков н.э. Y-ДНК: R-FT20255 Оба VK449 и VK259 делят 3 SNP с мужчиной из Швеции. Образует новую ветвь вниз R-FT20255 (Z18). Новая ветвь = R-FT22694 мтДНК: I2
Образец: VK260 / UK_Dorset-3735 Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK Возраст: Viking 10-11 веков н.э. Y-ДНК:
0t Q-BY77336 m
Образец: VK261 / UK_Dorset-3736 Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э. Y-ДНК: R-BY64643 Y-ДНК: R-BY64643 m
Образец: VK262 / UK_Dorset-3739 Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э. Y-ДНК: I-FT347811 FTDNA Комментарий: FT347811 2 SNP с американцем неизвестного происхождения.Образует новую ветвь вниз от Y6908 (Z140). В то же время была обнаружена новая ветвь, которая объединяет эту новую древнюю / американскую ветвь с установленной веткой I-FT274828. Новый древний путь = I-Y6908> I-FT273257> I-FT347811 мтДНК: J1c4
Образец: VK263 / UK_Dorset-3742 Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э. Y-ДНК: K-Z16372 m
Образец: VK264 / UK_Dorset-3744 Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания Возраст: Викинг 10-11 веков н.э.
Образец: VK267 / Sweden_Karda 21 Местоположение: Карда, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-L23 мтДНК: T2b4b
Образец: VK268 / Sweden_Karda 22 Местоположение: Карда, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-M269 мтДНК: K1c1
Образец: VK269 / Sweden_Karda 24 Местоположение: Карда, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-M269 mtDNA: h2e1a
Образец: VK273 / Россия_Гнездово 77-255 Местоположение: Гнездово, Россия Возраст: Викинг 10-11 вв. Н.э. Y-ДНК: R-BY61747 мтДНК: U5a2a1b1
Образец: VK274 / Дания_Kaargarden 391 Местоположение: Kaagården, Langeland, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-Ph4519 mtDNA: T2b-T152C!
Образец: VK275 / Дания_Kaargarden 217 Расположение: Kaagården, Langeland, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-BY74743 мтДНК: H
Образец: VK279 / Дания_Galgedil AX Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-Y10639 мтДНК: I4a
Образец: VK280 / Дания_Galgedil UO Местоположение: Галгедил, Фюн, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: I-Y3713 мтДНК: h21a
Образец: VK281 / Дания_Barse Grav A Местоположение: Борсе, Силенд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-FGC22153 Комментарий FTDNA: Разделяет I-Y5612 ( P109).Получено для 8, предков для 2. Новый путь = I-Y5612> I-Y5619 мтДНК: T2
Образец: VK282 / Дания_Stengade I, LMR c195 Местоположение: Stengade_I, Langeland, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-CTS1211 mtDNA: 8 h5a
Образец: VK286 / Дания_Bogovej Grav BJ Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-S10708 mtDNA: J1c-C16261
Образец: VK287 / Дания_Kaargarden Grav BS Местоположение: Kaagården, Langeland, Дания Возраст: Viking X век н.э. Y-ДНК: R-S22676 mtDNA: T2b
Образец: VK289 / Дания_Bodkergarden Grav H, sk 1 Местоположение: Bødkergarden, Langeland, Дания Возраст: Викинг 9 век н.э. Y-ДНК: R-U106 mtDNA:
8 J2b1a
Образец: VK290 / Дания_Kumle Hoje Grav O Местоположение: Kumle_høje, Langeland, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-FT264183 FTDNA Комментарий: Акционирует не менее SNP с мужчиной из Швеции, образуя новую ветвь ниже по течению R-FT263905 (U106).Новая ветка = R-FT264183. HG02545 остается в R-FT263905 мтДНК: I1a1
Образец: VK291 / Дания_Bodkergarden Grav D, sk 1 Местоположение: Bødkergarden, Langeland, Дания Возраст: Викинг, 9 век н.э. Y-ДНК: I-Y20861 mtDNA: U5a
Образец: VK292 / Дания_Bogovej Grav A.D. Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-M417 мтДНК:
028 J1c2c1 9
Образец: VK295 / Дания_Hessum sk 1 Местоположение: Хессум, Фюн, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: I-Y4738 мтДНК: T1a1
Образец: VK296 / Дания_Hundstrup Mose sk 1 Расположение: Hundstrup_Mose, Sealand, Дания Возраст: Ранний викинг 660-780 CE Y-ДНК: I-S7660 mtDNA: HV6
Образец: VK297 / Дания_Hundstrup Mose sk 2 Местоположение: Hundstrup_Mose, Sealand, Дания Возраст: Ранний викинг 670-830 CE Y-ДНК: I-Y4051 mtDNA:
0 J1c2h 9
Образец: VK301 / Дания_Ladby Grav 4 Местоположение: Ladby, Funen, Дания Возраст: Viking 640-890 CE Y-ДНК: I-FT105192 mtDNA: R0a2b
Образец: VK306 / Sweden_Skara 33 Расположение: Варнем, Скара, Швеция Возраст: Викинг 10-12 веков н.э. Y-ДНК: I-FT115400 FTDNA Комментарий: Имеет 3 мутации с мужчина из Швеции.Образует новую ветвь I-S19291. Новая ветка = I-FT115400. VK151 не имеет покрытия для 2 из этих мутаций мтДНК: h25a1
Образец: VK308 / Sweden_Skara 101 Расположение: Варнем, Скара, Швеция Возраст: Викинг 10-12 века н.э. Y-ДНК: R-BY33037 мтДНК: h2c
Образец: VK309 / Sweden_Skara 53 Местоположение: Варнем, Скара, Швеция Возраст: Викинг 10-12 веков н.э. Y-ДНК: R-YP6189 мтДНК: K1b1c
Образец: VK313 / Дания_Rantzausminde Grav 2 Местоположение: Ранцаусминде, Фюн, Дания Возраст: Викинг 850-900 CE Y-ДНК: R-JFS0009 мтДНК: h2b
Образец: VK315 / Дания_Bakkendrup Grav 16 Местоположение: Баккендруп, Силенд, Дания Возраст: Viking 850-900 CE Y-ДНК: I-Y98280 Комментарий FTDNA: Делится 1 SNP с мужчина из Нидерландов.Образует новую ветвь после I-Y37415 (P109). Новая ветвь = I-Y98280 мтДНК: T1a1b
Образец: VK316 / Дания_Hessum sk II Местоположение: Хессум, Фюн, Дания Возраст: Викинг 9-11 веков н.э. Y-ДНК: I-Y130659 Комментарий FTDNA: Сплит I- Y130594 (Z59). Получено для 1 предка для 6. Новый путь = I-Y130659> I-Y130594> I-Y130747. Древний образец STR_486 также принадлежит к этой группе, по I-Y130747 мтДНК: K1a4
Образец: VK317 / Дания_Kaargarden Grav BF99 Расположение: Kaagården, Langeland, Дания Возраст: Викинг 10 век н.э. Y-ДНК: J-BY62479 FTDNA Комментарий: 6-Splits J2 M67).Получено для 9, предков для 3. Новый путь = J-BY62479> J-BY72550 мтДНК: h3a2a1
Образец: VK320 / Дания_Bogovej Grav S Расположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-Y103013 Комментарий FTDNA: Делится 3 SNP с мужчина из Швеции. Образует новую ветвь вниз I-FT3562 (P109). Новая ветвь = I-Y103013 мтДНК: U5a1a1
Образец: VK323 / Дания_Рибе 2 Местоположение: Рибе, Ютландия, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: R-S10185 мтДНК: K2a6
Образец: VK324 / Дания_Ribe 3 Расположение: Рибе, Ютландия, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: R-BY16590 FTDNA Комментарий: Сплит R-BY16590 (L47).Получено для 7, предков для 3. Новый путь = R-S9742> R-BY16950 мтДНК: N1a1a1a2
Образец: VK326 / Дания_Рибе 5 Местоположение: Рибе, Ютландия, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: R-Y52895 mtDNA: U5b1-T16189C! — T16192C!
Образец: VK327 / Дания_Рибе 6 Местоположение: Рибе, Ютландия, Дания Возраст: Викинг 9-11 века н.э. Y-ДНК: I-BY463 мтДНК: H6a1a5
Образец: VK329 / Дания_Ribe 8 Местоположение: Рибе, Ютландия, Дания Возраст: Викинги 9-11 веков н.э. Y-ДНК: R-S18894 мтДНК: h4-T152C!
Образец: VK332 / Oland_1088 Местоположение: Эланд, Швеция Возраст: Viking 858 ± 68 CE Y-ДНК: I-S8522 FTDNA Комментарий: Возможно, ниже I-BY195155.Имеет одну мутацию C> T с образцом BY195155 * мтДНК: T2b24
Образец: VK333 / Oland_1028 Местоположение: Эланд, Швеция Возраст: Viking 885 ± 69 CE Y-ДНК: R-Z29034 mtDNA: h3a2a1
Образец: VK335 / Oland_1068 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 веков н. Э. Y-ДНК: R-BY39347 FTDNA Комментарий: Делится 8 SNP с мужчиной Франция.Образует новую ветвь вниз по R-BY39347 (U152). Новая ветвь = R-FT304388 мтДНК: K1b2a3
Образец: VK336 / Oland_1075 Местоположение: Эланд, Швеция Возраст: Viking 853 ± 67 CE Y-ДНК: R-BY106906 мтДНК: K2a3a
Образец: VK337 / Oland_1064 Местоположение: Эланд, Швеция Возраст: Viking 858 ± 68 CE Y-ДНК: I-BY31739 FTDNA Комментарий: Возможный Z140 mtDNA1b: 9003a U5a
Образец: VK338 / Дания_Bogovej Grav BV Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-A6707 мтДНК: W3a1
Образец: VK342 / Oland_1016 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: I-BY78615 FTDNA Комментарий: Имеет 2 SNP с мужчиной из Финляндия.Образует новую ветвь вниз I2-Y23710 (L801). Новая ветвь = I-BY78615 мтДНК: h3a1
Образец: VK343 / Oland_1021 Расположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: I-Y7232 мтДНК: h4h
Образец: VK344 / Oland_1030 Расположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-BY32357 mtDNA: J1c2t
Образец: VK345 / Oland_1045 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: R-FT148754 FTDNA Комментарий: Сплит R-FT148754 (DF63) .Получено для 8, предков для 6. Новый путь = R-FT148796> R-FT148754 мтДНК: h5a1
Образец: VK346 / Oland_1057 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 века н.э. Y-ДНК: J-Z8424 мтДНК: h3a2b
Образец: VK348 / Oland_1067 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: I-Z171 мтДНК: T2b28
Образец: VK349 / Oland_1073 Местоположение: Оланд, Швеция Возраст: Viking 829 ± 57 CE Y-ДНК: R-BY166065 FTDNA Комментарий: Делится 2 SNP с мужчиной из Англии .Образует ответвление вниз от R-BY166065 (L1066). Новая ветвь = R-BY167052 мтДНК: h2e2a
Образец: VK352 / Oland_1012 Местоположение: Эланд, Швеция Возраст: Викинг 9-11 веков н.э. Y-ДНК: I-FGC35755 FTDNA Комментарий: Возможно, образует ветвь вниз I -Y15295. 2 возможные мутации G> A с образцом I-Y15295 * мтДНК: H64
Образец: VK354 / Oland_1026 Местоположение: Эланд, Швеция Возраст: Viking 986 ± 38 CE Y-ДНК: R-S6752 мтДНК: h3a1
Образец: VK355 / Oland_1046 Местоположение: Эланд, Швеция Возраст: Викинг 847 ± 65 CE Y-ДНК: L-L595 FTDNA Комментарий: Присоединяется к 2 другим древним на этой редкой ветви .ASH087 и I2923 мтДНК: U5b1b1a
Образец: VK357 / Oland_1097 Местоположение: Эланд, Швеция Возраст: Viking 1053 ± 60 CE Y-ДНК: I-FT49567 FTDNA Комментарий: Делится 4 SNP с мужчиной из Англии . Образует новую ветвь I-A5952 (Z140). Новая ветвь = I-FT49567 мтДНК: J2b1a
Образец: VK362 / Дания_Bogovej LMR 12077 Расположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: E-CTS5856 FTDNA Комментарий:
3 Возможно 90 E-Z мтДНК: V7b
Образец: VK363 / Дания_Bogovej BT Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-BY198083 Комментарий FTDNA: Делится 2 SNP с мужчиной из Швейцарии.Образует новую ветвь I-A1472 (Z140). Новая ветвь = I-BY198083 мтДНК: U4b1a1a1
Образец: VK365 / Дания_Bogovej BS Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: R-BY34800 мтДНК: U8a2
Образец: VK367 / Дания_Bogovej D Местоположение: Богёвей, Лангеланд, Дания Возраст: Викинг 10 век н.э. Y-ДНК: I-BY67827 Комментарий FTDNA: VK506 и VK19967 9 Внутренние отделы — SCP Foundation
- Административный отдел
- Департамент иностранных дел (DEA)
- Отдел инженерно-технического обслуживания
- Комитет по этике
- Разведывательное управление
- Отдел внутренней безопасности
- Отдел логистики
- Производственный отдел
- Лечебное отделение
- Мобильные оперативные группы
- Научный отдел
- Отдел безопасности
- Департамент внутреннего трибунала
- Центральные базы данных мутаций и SNP
Армирование ленточного фундамента
Бетон – это основная составляющая ленточного фундамента. По своим свойствам он не имеет большую прочность и при малейшей сейсмической активности либо механическом воздействии даст трещину. Чтоб предотвратить разрушение самой главной части здания – фундамента, строители уже более двух веков используют технологию армирования бетона. Таким образом, с помощью арматурных прутьев создается основание с высокой прочностью и эластичностью. Довольно часто на фундамент воздействует неравномерная нагрузка, которая может объясняться разной структурой грунта либо существенным отличием массы определенных частей построенного здания. Под таким давлением верхняя часть фундамента сжимается, а нижняя растягивается. Армированный же слой противостоит этому растяжению, сохраняя прочность железобетонного изделия на протяжении 150 лет. Армирование ленточного фундамента производится в несколько этапов. Рассмотрим их более подробно.
Армирование фундамента арматурой
Для возведения ленточного фундамента используют арматурные прутья разных диаметров от 6-8 мм до 10-14 мм. Металлический каркас фундамента соединяется с помощью проволоки, данный процесс называется вязка арматуры. Чтоб правильно сделать расчет арматуры для фундамента необходимо учитывать следующие моменты:
Металлический каркас фундамента соединяется с помощью проволоки, данный процесс называется вязка арматуры. Чтоб правильно сделать расчет арматуры для фундамента необходимо учитывать следующие моменты:
-
Элементы каркаса, которые будут монтироваться горизонтально должны иметь максимальную прочность. Их диаметр выбирают с учетом качества грунта. Чем больше структура почвы отличатся по всему периметру, тем толще необходимо использовать металлические прутья. Чаще всего их диаметр колеблется в пределах 10-14 мм. Поверхность продольных прутьев должна иметь ребра для лучшей сцепки с бетоном. Для поперечных элементов можно использовать тоненькие и гладкие прутья (6-8мм). Они не подвергаются сильной нагрузке, при этом значительно меньше стоят. -
Продольная арматура, которая укладывается по всему периметру фундамента, должна находиться на расстоянии 5 см от стен опалубки, дна траншеи, а так же от верхней части фундамента. Таким образом, бетон, покрывая все элементы каркаса, защитит их от коррозии.
-
Учитывая предыдущую рекомендацию, для ленты фундамента шириной 40 см необходимо использовать армированный каркас шириной 30 см. Высота его может колебаться в пределах 10-30 см (в зависимости от глубины траншеи, предполагаемой нагрузки и структуры почвы). Расстояние между поперечными элементами так же варьируется в пределах 10-30 см.
С глубиной траншеи не более 1,2 м используют три пары продольных прутьев. Соединяются они между собой двумя тонкими прутьями. Скрепление каркаса с помощью сварки не рекомендуется проводить, так как от воздействия высокой температуры металл теряет свою крепость. Для обвязки арматуры проволокой можно использовать специальный строительный крючок. Самым проблемным моментом при создании каркаса считаются углы. В предыдущей статье мы рассмотрели способы рытья котлованов.
Армирование углов
Углы ленточного фундамента подвергаются сильным нагрузкам.
При изготовлении каркаса в этих местах необходимо создать высокую прочность.
Обычное скрещение арматурных прутьев не создаст единой крепкой конструкции, что приведет к образованию трещин.
По правильной технологии армирования ленточного фундамента, прутья в угловых местах необходимо сгибать.
СНиП армирования фундаментов
Очень важно соблюдать все нюансы армирования ленточного фундамента. Это позволит построить долговечное здание с основанием, устойчивым к различным механическим нагрузкам, сейсмической активности и другим неблагоприятным факторам. Более детальную инструкцию армирования фундамента можно прочитать в специальном пособии к СНиП 2.03.01-84 «Бетонные и железобетонные конструкции» и СНиП 2.02.01-83 «Основания зданий и сооружений». Конечно, там все описано техническим языком. Несмотря на это данная инструкция содержит всю необходимую информацию по строительству ленточного фундамента.
Видео об армировании ростверка
Армирование ленточного фундамента СНИП — bmi-rus.ru
Схема армирования ленточного фундамента по СНиП
Тяжесть любого здания передается на грунт через фундамент. Фундамент не позволяет строению деформироваться или смещаться под отрицательным воздействием почвы и климатических условий. Эта важная конструкция может быть линейной, столбчатой, плитной (плавающей), свайной. Первые три вида требуют использования бетонной смеси и ее армирования.
Для чего нужно армировать фундамент
Фундамент чаще всего деформируется из-за неравномерной нагрузки или пучения грунта под воздействием низких температур. Если конструкция состоит из бетона, то следует учитывать его характеристики: высокие показатели прочности на сжатие и низкую прочность на разрыв. Для компенсации последнего качества используется схема каркаса, которая монтируется из металлических прутьев для армирования.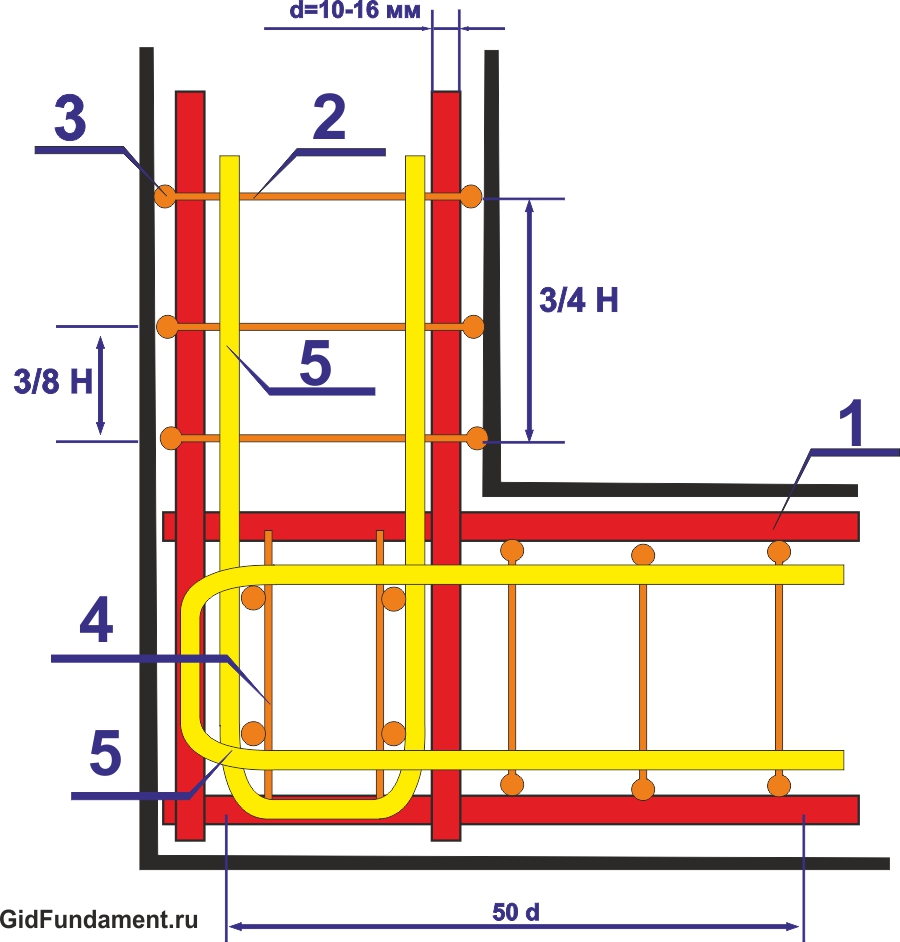 Сталь обладает более высокой устойчивостью к растяжению, что помогает фундаменту выдерживать повышенные нагрузки.
Сталь обладает более высокой устойчивостью к растяжению, что помогает фундаменту выдерживать повышенные нагрузки.
Верхняя часть конструкции фундамента под весом здания сжимается, нижняя растягивается при замерзании грунта, вследствие чего в области растяжения могут появиться трещины. Поэтому арматура укладывается в нижней и верхней части фундамента. В армированном бетоне цементный раствор сопротивляется сжатию, металл — процессу растяжения. Укладывать прутья посередине нет смысла, так как там повышенной нагрузки не наблюдается.
При возведении фундамента особое внимание необходимо уделить тем частям конструкции, которые выделяются на пристройки и эркеры. Для армирования бетона в этих областях используются согнутые под определенным углом прутья на примыкающие стены. Металл не должен выступать за опалубку или уходить в грунт, расстояние между прутьями не должно превышать 5 см. Для соединения можно использовать только проволоку (но не сварку). Форма каркаса из арматурных прутьев должна быть квадратной (прямоугольной).
Форма каркаса из арматурных прутьев должна быть квадратной (прямоугольной).
Требования СНиП к монтажу арматуры
Общие схемы и требования к возведению конструкций с использованием бетона (железобетона) определены в СНиП 52−01−2003. Данный документ содержит правила расчета склонности железобетона к деформациям, его способности к образованию трещин, показателей прочности, требования к размерам и формам конструкции:
- при возведении фундаментов можно использовать только арматуру, соответствующую стандартам, с сертификатом качества, определенную в проектной документации;
- прутья сцепляются так, чтобы полностью исключить возможность их смещения во время заливки бетона;
- если для армирования ленточного фундамента используются сварные каркасы или сетки, то при их изготовлении разрешается применять такой способ сварки, который не допускает деформирования;
- радиус изгиба арматурных прутьев должен соответствовать затребованному в проекте;
- механические стыки арматуры по прочности не должны уступать прочности основного материала;
- расстояние между вертикальными стержнями зависит от их диаметра, вида заполнителя бетонной смеси, расположения в каркасе, метода заливки бетона, но не допускается шаг меньше, чем 25 см;
- расстояние между продольными прутьями не должно превышать 40 см;
- расстояние между прутьями, установленными поперечно, не должно превышать 30 см.

Для вертикального армирования используются прутья с диаметром 10−12 мм с ребристой поверхностью. Для продольного расположения диаметр арматуры не должен быть меньше, чем 10 мм и больше, чем 32 мм. Для поперечного размещения используется арматура с диаметром от 6 до 8 мм.
Как правильно армировать ленточный фундамент
Перед тем как заливать ленточный конструкцию, необходимо ее армировать при помощи металлической арматуры. Ленточный фундамент — полоса из железобетона по всему периметру дома, заложенная под наружными и внутренними стенами. Толщина конструкции зависит от материала стен и их толщины.
Мелкозаглубленные фундаменты (глубина от 50 до 70 см) возводятся на пучинистых почвах для строений из бревна или бруса, а также каменных домов с площадью не более чем 6×6 м. Заглубленные фундаменты возводятся при строительстве больших и тяжелых домов с цоколями, подвалами и гаражами. Глубина заглубленной конструкции — на 20−30 см ниже, чем уровень замерзания грунта.
Количество арматурных сеток зависит от вида фундамента. Для конструкции глубиной 50 см и шириной 40 см шаг между продольными прутьями может быть 10−15 см. Если высота конструкции около метра, то между горизонтальными прутьями с ребрами и диаметром 10−16 мм должно быть 30−40 см. Вертикальная арматура (гладкие прутья с диаметром 6−8 мм) устанавливается, если высота фундамента больше, чем 15 см. В любом случае арматура для ленточного фундамента должна иметь структуру жесткой рамы прямоугольного или квадратного сечения.
Особая разновидность ленточного фундамента — конструкция с пенополистирольной несъемной опалубкой в виде листов или пустотелых блоков, которые также подвергаются армированию. Подобная опалубка собирается просто, а после заливки бетонной смеси она не требует разборки.
Диаметр прутков должен быть примерно 0,1% от площади поперечного сечения основы будущего здания. Армирование в пенополистирольной опалубке производится горизонтально и вертикально. Шаг между горизонтальными элементами согласно СНиП — 50 см. Если монтируется этот вид ленточного фундамента, то специалисты советуют дополнить его гидроизоляцией. Недавно рынок стал предлагать пенополистирольную опалубку с арматурой, что позволяет избежать необходимости в ее вязке.
Шаг между горизонтальными элементами согласно СНиП — 50 см. Если монтируется этот вид ленточного фундамента, то специалисты советуют дополнить его гидроизоляцией. Недавно рынок стал предлагать пенополистирольную опалубку с арматурой, что позволяет избежать необходимости в ее вязке.
Как армировать фундамент столбчатой конструкции
Столбчатый фундамент — это вкопанные в грунт столбы различной формы, расположенные в местах, где пересекаются стены, а также в пролетах. Их нижнюю часть называют основанием, верхнюю — оголовком. Оголовок должен быть идеально ровным, располагаться от 40 до 50 см над грунтом (на него возводятся стены). Этот вид фундамента можно использовать практически в любом грунте (кроме пучинистого), он менее затратный, чем ленточный, легко монтируется собственными силами.
Столбы для фундамента можно брать круглые, квадратные или прямоугольные. Опалубка строится:
- из досок толщиной не менее 4 см,
- ДСП,
- фанеры,
- железа.

При круглом сечении вместо опалубки можно использовать трубы длиной 2−2,5 м, с диаметром 10−20 см. Скважины круглой формы высверливаются ручным буром. Для армирования достаточно двух вертикальных прутьев с ребрами, перевязанных в трех или четырех местах монтажной проволокой.
Столбы квадратной формы можно сделать не только с одинаковым, но и с различным сечением на концах (в виде ровного параллелепипеда или с расширенным основанием). Расширение увеличивает показатели несущей способности и сопротивляемости деформациям при промерзании грунта. Для установки столбов квадратной или прямоугольной формы роются ямы и монтируется опалубка, задающая форму столба. Перед заливкой бетонной смеси на дно устанавливается гидроизоляция и монтируется арматура из вертикальных прутьев, перевязанных проволокой.
Угол стыковки арматуры необязательно должен быть 90 градусов. Главное, чтобы не нарушалась общая картина армирования фундамента, схема, которая соответствует проекту. Армирование углов ленточного фундамента производится аналогично армированию основной конструкции.
Армирование углов ленточного фундамента производится аналогично армированию основной конструкции.
Для заливки можно использовать стандартную бетонную смесь (марка В25) или добавить в нее бутовый камень или плитняк средних размеров. Смесь заливается постепенно, примерно по 20 см, чтобы предотвратить скопление воздуха. После затвердения бетона опалубка демонтируется, столбы засыпаются грунтом.
Армирование плитной конструкции фундамента
Плитная (плавающая) конструкция фундамента — это цельная плита из железобетона, толщина которой 10 см или более, уложенная на подушку из песка и гравия и расположенная по всей площади здания. Этот вид конструкции фундамента бывает двух видов:
Для мелкозаглубленной конструкции достаточно снять верхний слой грунта и заменить его подушкой из песка и гравия. При установке заглубленного фундамента требуется рытье достаточно глубокого котлована, поэтому подобные конструкции сооружаются при возведении домов с цоколями или подвалами.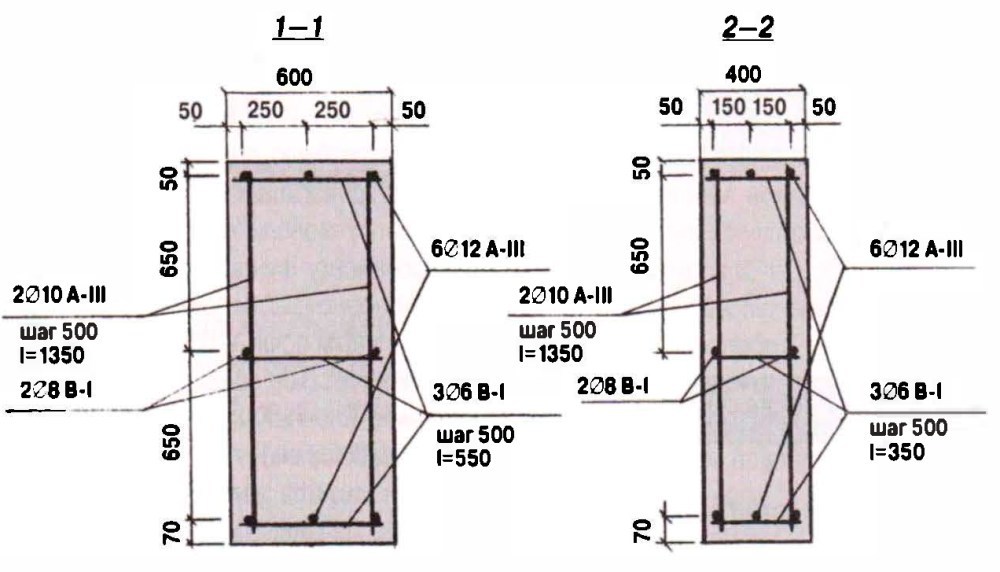
На подушку из гравия и песка укладывается гидроизоляционный материал и монтируется опалубка. Потом создается арматурный короб, состоящий из нижней и верхней сетки, которые связаны между собой. Используются прутья с ребрами и диаметром от 12 до 16 мм, расположенные на расстоянии 20 см друг от друга. Арматурные прутья можно заменить вязаной сеткой или каркасом, соединенным резьбовыми соединениями. Сетки можно укладывать в двух, трех или четырех плоскостях. Независимо от вида арматуры, необходимо монтировать ее так, чтобы верхняя часть плиты после заливки бетона была гладкой.
Построить фундамент из бетона можно и своими руками, если все правильно рассчитать и выбрать соответствующую марку бетона и арматуру. Для ленточной, столбчатой и мелкозаглубленной плитной конструкции даже земельные работы можно выполнить вручную. Трудности могут возникнуть только с заглубленным плитным фундаментом, требующим рытья глубокого котлована и большого объема бетона.
Снип фундаменты ленточные
Как в промышленном, так и в индивидуальном строительстве самым надежным фундаментом считается армированный ленточный. Это основание из бетона, которое формируется в траншее определенной глубины и ширины, с армированием металлическим каркасом и последующей заливкой раствором. Любой фундамент испытывает всевозможные нагрузки – на растяжение и сжатие, на изгиб и излом, поэтому к таким конструкциям предъявляются жесткие требования по различным параметрам, описанные в соответствующих ГОСТ и СНиП. Так как требований достаточно много, запоминать их не
Это основание из бетона, которое формируется в траншее определенной глубины и ширины, с армированием металлическим каркасом и последующей заливкой раствором. Любой фундамент испытывает всевозможные нагрузки – на растяжение и сжатие, на изгиб и излом, поэтому к таким конструкциям предъявляются жесткие требования по различным параметрам, описанные в соответствующих ГОСТ и СНиП. Так как требований достаточно много, запоминать их не
Перечень основных документов для строительства армированных оснований
Схема армирования и технология строительства основания
Армирование бетонной формы основания проводится в два яруса – верхним и нижним рядами арматуры с поперечным и продольным усилением дополнительными прутьями. Для формирования прочного, но гибкого армокаркаса применяют арматурные прутья категории А III – это стальной профиль круглого сечения Ø 10-16 мм, имеющий два продольных ребра жесткости и поперечные грани, отлитые по спирали.
При общей высоте основания ≥ 0,15 м в каркас необходимо встраивать вертикальные стержни арматуры, что делается методом связывания при помощи мягкой вязальной проволоки (СНиП 52-01-2003 и СП 52-101-2003). Для вертикального усиления каркаса применяют арматуру класса А I – это гладкая арматура Ø 6-8 мм. Чтобы компенсировать продольные нагрузки в теле бетонного ленточного фундамента, каркас усиливается поперечной арматурой, которая предотвращает образование микротрещин и скрепляет друг с другом продольные ярусы армирующего каркаса основания.
Для вертикального усиления каркаса применяют арматуру класса А I – это гладкая арматура Ø 6-8 мм. Чтобы компенсировать продольные нагрузки в теле бетонного ленточного фундамента, каркас усиливается поперечной арматурой, которая предотвращает образование микротрещин и скрепляет друг с другом продольные ярусы армирующего каркаса основания.
Онлайн калькулятор для расчета арматуры
Согласно указанным СНиП, вертикальная и поперечная арматура связывается в единую конструкцию стальными хомутами, расстояние между которыми соблюдается как 3/8 от высоты ленточного фундамента, и должно быть ≥ 0,25 м.
Также армирующий каркас в соответствии со снип фундаменты ленточные не должен собираться из поврежденных или ржавых стержней – арматура должна быть ровной и порезанной по расчетным размерам. Отдельные арматурные прутья также соединяются между собой при помощи мягкой или отожженной вязальной проволоки и вязального крючка. Применять сварочное оборудование разрешено только для соединения прутьев с мариковкой «С».
Армирование ленточных оснований
Правила связывания армирующего каркаса должны соблюдаться неукоснительно, иначе не получится добиться требуемой жесткости каркаса. Связывание углов и присоединений каркаса предотвращает разрушающее воздействие локальных нагрузок на фундамент. Для угловых примыканий используются арматурные прутья класса А III. Основные рекомендации при соединении углов армокаркаса:
- Прут необходимо согнуть в таким образом, чтобы один его конец входил в стену основания, второй конец входил в противоположную стену;
- Запускать стержень арматуры на противоположную стену следует на длину сорока диаметров прута;
- Не разрешается применять простое связывание пересечений арматуры без из усиления дополнительными вертикальными и поперечными отрезками арматуры;
- При длине прута, не позволяющей загнуть его на противоположную стену фундамента, арматура соединяется Г-образными металлическими профилями;
- Шаг между соединительными хомутами выбирается в два раза короче, чем в ленте.

Схема связывания арматуры
Заливка бетона в траншею
Требования к заливке бетонного раствора в фундамент предъявляются во многих документах – ТСН 50-302-2004, ВСН 29-85, ГОСТ 13580-85, СП 63.13330.2013, СП 52-101-2003, СНиП 52-01-2003, СП 22.13330.2011, ГОСТ Р 54257-201, и других. Раствор заливается в ограниченную опалубкой траншею послойно, с толщиной пластов 0,20-0,25 м. Укладка раствора ведется в одном направлении, но при большой ширине ленты допускается заливка наклонных слоев под углом ≤ 30 0 .
» alt=»»>
После заливки одного слоя и распределения раствора весь бетон необходимо уплотнить вибратором или ручным штыкованием лопатой или ломом, чтобы высвободить находящийся в растворе воздух, который ослабляет бетон и делает его более уязвимым для разрушения при воздействии разновекторных нагрузок. Следующий шаг – укладка верхнего слоя раствора. Если лента фундамента широкая и глубокая, то необходимо сделать холодный шов. Если предыдущий слой бетона схватился и затвердел, то его поверхность перед укладкой следующего пласта раствора необходимо очистить и обезжирить, а затем просушить потоком теплого воздуха. Очистка холодного рабочего шва обязательна, так как заливка на грязную поверхность верхнего слоя бетона разрушит монолитную конструкцию основания из-за находящейся между пластами раствора грязи и цементной пленки. Основные положения по формированию ленты фундамента регламентированы в указанных выше документах.
Очистка холодного рабочего шва обязательна, так как заливка на грязную поверхность верхнего слоя бетона разрушит монолитную конструкцию основания из-за находящейся между пластами раствора грязи и цементной пленки. Основные положения по формированию ленты фундамента регламентированы в указанных выше документах.
Выдержка из СНиП
Очищают поверхность бетона от цементной пленки металлической щеткой (при прочности бетона ≥ 1,5 МПа), фрезерованием (при прочности бетона ≥ 5 МПа), пескоструйкой (при прочности бетона ≥ 5Мпа) или промывкой струей воды (при прочности бетона ≥ 0,3 МПа). Самый дешевый метод – очистка водой, и этот пункт также влияет на общую стоимость ленточного фундамента.
Холодный рабочий шов расположен в теле основания не только горизонтально, но и вертикально и перпендикулярно относительно осей балок, стен, колонн и плит. Отсекают рабочий шов щитом из досок или фанеры, а для свободного прохождения арматуры в нем проделываются отверстия соответствующего диаметра под прутья каркаса.
Перед тем, как залить ленточный фундамент снип, выжидают определенное время для достижения прочности бетона в предыдущем слое не менее 1,5 МПа. Первые 3-5 суток незатвердевший слой защищают от осадков и солнечных лучей, мороза или жары. Механические повреждения бетона в этот период также недопустимы, пока прочность бетона не увеличится до 1,5МПа.
Общие положения СНиП при проектировании фундаментов
Калькулятор вес арматуру
Как проверяется прочность бетона
Прочность материалов – это способность сопротивляться разрушительным воздействиям под влиянием внутреннего напряжения материала, возникающего под давлением сил извне или из-за других факторов (усадка, влажность, температура, и т.д.).
Свойства прочности материала рассчитываются несколькими методами:
- Метод стандартных образцов;
- Метод исследования выбуренного керна;
- Метод неразрушающего контроля, который считается самым дешевым и действенным.
Проверка прочности бетона
Расчет материалов
Количество и вес арматурных стержней, которое потребуется для конструирования армирующего каркаса, рассчитывается по габаритам ленты фундамента.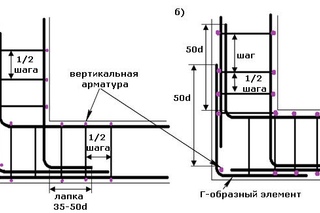 При ширине ленты 0,4 м рекомендуется использовать четыре продольных прута – по два сверху и снизу. В качестве примера можно рассмотреть формирование каркаса 6 х 6 м для ленточного основания дома.
При ширине ленты 0,4 м рекомендуется использовать четыре продольных прута – по два сверху и снизу. В качестве примера можно рассмотреть формирование каркаса 6 х 6 м для ленточного основания дома.
При четырехрядной укладке понадобится 24 м арматуры на один ряд, для всего каркаса – 96 м. Вертикальные и поперечные гладкие стержни армирования для фундамента ленты шириной 30 см и высотой 190 см: для каждой точки пересечения прутьев при шаге 0,05 м от верхней части фундамента понадобится (30 – 5 – 5) х 2 + (190 – 5 – 5) х 2 = 0,40 м. Расстояние между стальными хомутами 50 см, количество хомутов: 24 / 0,5 + 1 = 49 единиц.
» alt=»»>
Общий метраж армирующих прутьев для формирования каркаса по вертикали составит 4 х 49 = 196 м. Каждое место связывания – это четыре пересечения, поэтому расход вязальной проволоки для каждого соединения – восемь отрезков по 30-40 см. Общий метраж составит: 0,3 х 8 х 49 = 117,6 метра.
Расчет арматуры
Ленточный фундамент по монолитному типу формируется в виде прямоугольника или квадрата. Армирующий каркас формируется в результате нескольких последовательных операций:
Армирующий каркас формируется в результате нескольких последовательных операций:
- Дно траншеи прерывисто укладывается кирпичами высотой в четверть кирпича, чтобы можно было залить раствором промежуток между каркасом и подошвой фундамента;
- Под стойки арматурного каркаса делается шаблон, по нему нарезаются отрезки арматуры нужного размера;
- На слой кирпича кладутся продольные прутья армирующего каркаса. Если прутья короткие, их связывают с нахлестом ≥ 0,2 м;
- Горизонтальные гладкие прутья связываются в каркасе с продольной арматурой с шагом 0,5 м;
- По углам ячеек из арматуры привязываются вертикальные гладкие стержни длиной на 10 см короче высоты основания;
- Продольная арматура привязывается к вертикальным стержням;
- К углам, которые получились в результате этих операций, привязываются поперечные верхние стержни.
Заливка ленточного основания бетоном
Требования СНиП
По поводу строительства фундамента ленточного типа: существует документ СНиП 52-01-2003, регламентирующий расстояния между прутьями каркаса, в частности, шаг между горизонтальными гранями армокаркаса и шаг между поперечными прутьями. Это расстояние зависит от:
Это расстояние зависит от:
- Диаметра арматуры;
- Фракции бетонного заполнителя;
- Ориентирования каркаса относительно бетонирования;
- Метода заливки раствора в опалубку;
- Типа уплотнения раствора.
Требования определяют, что шаг продольного армирования регламентируется как H = ≤ 40 см и ≥ 25 см. Расстояние между поперечными прутьями арматуры определяется как 1/2 высоты сечения ленты, но не больше, чем 0,3 м.
Диаметр армирования зависит от общего метража продольного армирования фундамента и предполагается ≥ 0,1% площади сечения ленты. На практике это означает, что для бетонного основания высотой 100 см при ширине ленты 50 см площадь сечения будет равняться 500 мм 2 .
Размеры фундаментной ленты согласно СНиП
МЗЛФ (мелкозаглубленный фундамент) отличается от заглубленного высотой бетонной ленты, поэтому глубокозаглубленные в фундаменты закладывается более развитая структура каркаса, боковых бетонных стенок и подошвы. Из-за большой глубины такого основания существуют рекомендации от профессионалов: для лент глубиной ≤ 1 м армируется только подошва фундамента, а в глубокозаглубленных основаниях армируется также оболочка и днище.
Из-за большой глубины такого основания существуют рекомендации от профессионалов: для лент глубиной ≤ 1 м армируется только подошва фундамента, а в глубокозаглубленных основаниях армируется также оболочка и днище.
Дополнительное усиление армирующего каркаса в МЗЛФ проводится армирующей металлической сеткой из прутьев Ø 4 мм с размером ячеек 10 х 10 см. Любой тип армирования намного повышает прочность и жесткость конструкции, а также усиливает сопротивление опорной части ленты боковым и сжимающим нагрузкам.
Сама методика армирования бетонного основания не представляется сложной, и ее можно провести самостоятельно, что позволит не только усилить основание дома, но и добиться значительного снижения стоимости строительства.
» alt=»»>
Создание ленточного фундамента по нормам СНИП
Правила и нормы строительных работ прописываются в таких документах, как Снип – это сборка всех необходимых требований к совершению постройки архитектурного объекта. Если вы решили возводить сооружение, то должны строго придерживаться прописанных положений свода. Помимо правил, Снип, содержит информацию об определениях работ и составляющих их элементов. Так, исходя из документов, ленточный фундамент – это основание постройки, которое предназначено для возведения на непромерзающих глиняных почвах. В нашей статье мы и поговорим о требованиях к данному объекту дома.
Помимо правил, Снип, содержит информацию об определениях работ и составляющих их элементов. Так, исходя из документов, ленточный фундамент – это основание постройки, которое предназначено для возведения на непромерзающих глиняных почвах. В нашей статье мы и поговорим о требованиях к данному объекту дома.
Определение по Снип
Ленточные основания способны переносить достаточно высокое давление, благодаря чему могут применяться при строительных задачах для массивных каменных зданий. Его огромный плюс – это не склонность к различным видам деформаций. Снип свидетельствует о принадлежности этого фундамента для архитекторских проектов, которые имеют подвальное или цокольное пространство.
Ленточный фундамент располагается ниже уровня промерзания почвы, потому что практически все типы грунта разбухают после зимнего сезона. Если же не выдержать эту норму, то к весне основание может поплыть.
Внимание! Всю информацию об уровне промерзания грунтов по всей территории России можно отыскать в Снип.
Толщина стен и вид почвы становятся единственными факторами при расчетах размеров фундаментов. Поэтому и его расположение может быть как на большой глубине, так и на поверхности. Прежде всего, на это влияет еще и материал, из которого создано основание.
По уровню нагрузки выделяют такие виды ленточных фундаментов:
- Заглубленный вид , который предназначен для массивных построек на мягкой почве;
- Мелко заглубленный фундамент , который обычно применяется для мелких построек, заборов, а также деревянных домов.
Внимание! Независимо от показателей глубины, постройку необходимо выполнять согласно требованиям и нормам Снип.
Нормативно-законодательная база
Если вы собираетесь строить ленточный фундамент, то данные ГОСТа и Снип вам обязательно пригодятся:
- Основы строительных работ по созданию фундаментов из железобетонных плит записаны в Гост 13580-85;
- Все нормы к фундаментам постройки сведены в Снип 2.02.
 01.83;
01.83; - Документ о несущих и ограждающих постройках называется Снип 3.03.01-87;
- Все нормы и требования к возведению фундаментов и других земельных зданиях занесены в Снип 3.02.01-87.
Если вы будете следовать данной нормативно-законодательной базе, то можете не переживать надежности вашего строительного проекта.
Нормы СНИП к арматуре
Снип 52-01-2003 содержит все основные схемы и требования к конструкции постройки из железобетона. Также, в нем зафиксированы основные виды деформаций, показатели прочности, требования к размерам:
- При выполнении строительных работ по возведению фундамента необходимо использовать арматурное устройство с наличием сертификата качества;
- Прутья нужно скреплять плотно, чтоб исключить их смещение при заливке раствором;
- При использовании сварных деталей арматуры разрешено применять метод сварки, который не вызывает изменение форм;
- Изгиб прутьев должен иметь радиус, который идентичен, зафиксированной его величине в строительном плане;
- Устройство должно иметь стыки, которые должны совпадать с главным материалом по прочности;
- Дистанция между вертикальными стержнями ленточного основания определяется согласно их диаметру.
 Учитываются также виды заполнителя смеси.
Учитываются также виды заполнителя смеси. - Шаг, при заливке должен быть больше 25 см;
- Отрезок между двумя продольными прутьями – не больше 40 см;
- Расстояние между поперечными прутьями – не больше 30 см;
При вертикальном армировании используются элементы диаметром 12 см, а для продольного – от 10 до 32 см. Стоит отметить, что при поперечном процессе величина должна иметь показатель 7 см.
Этапы строительства ленточного фундамента по СНИП
Данное устройство состоит из бетона, который проходит армирование и после этого, заливается в опалубку, тем самым образуя монолитный комплекс. Существуют разнообразные виды возведения ленточного основания, но мы рассмотрим наиболее оптимальную и простую схему процесса.
Составление проекта
На этом этапе производится расчет всех необходимых величин, а именно:
- Глубина;
- Ширина;
- Выбор материала;
- Установление уровня промерзания почвы;
- Другие параметры грунтов.

Устройство должно проходить по всему периметру постройки, поэтому эти данные играют огромную роль в строительных работах.
Важно! Если постройка имеет форму – не квадрат, то установка ленты будет более сложной.
Выполнение разметки
После окончания проекта, необходимо расставить отметки будущего фундамента. Это совершается таким образом: колышки расставляются по периметру и обтягиваются шнуром по внешнему и внутреннему пространству. Когда вы возводите здание на мягком грунте, то траншея должна быть немного шире. Это необходимо для использования опалубки при выполнении работ. Также необходимо предусмотреть подушку 10 см, которая засыпается песком.
Земляные работы
На этом этапе происходит выполнение траншеи. Глубина должна быть идентичной величине фундамента, но иметь запас в 30 см для подушки. Для выполнения данной задачи лучше использовать натянутую веревку, чтоб не сбиваться от разметок. При земельном рытье учитывайте особенности почвы. Так, например, для твердых грунтов лучше делать вертикальные стены для канав.
Так, например, для твердых грунтов лучше делать вертикальные стены для канав.
Важно! Если на вашем участке сыпучие грунты, то габариты траншеи должны быть больше, чем прописаны в проекте/
Установка опалубки
Устройство опалубки возводится снаружи основания дома, то есть должна ширина досок соответствовать проектной величине. Процесс монтажа достаточно прост и выполняется примерно так же, как с деревянными щитками. По окончании ее возведения необходимо засыпать речным песком дно канала и хорошенько утрамбовать. Это и называется подушкой. Если добавить щебень и залить бетоном, то образуем подошву постройки.
Армирование
Следующим этапом необходимо выполнить армирование. Для этого пригодятся прутья диаметром 12 см и проволока, которой будет скрепляться конструкция. Детали арматуры по вертикали должны иметь расстояние от фундамента 10 см и связываться проволокой по всем направлениям. В конце работы мы получим пояс, который и будет выполнять армирование.
Выполнение заливки раствором бетона
При выполнении заливки одновременно на всех участках, необходимо использовать несколько машин для замеса раствора и перемешивать вылитый бетон ломом, чтоб избежать образования пустых пространств.
Если же все работы выполняются постепенно, то бетон будет и так ложиться равномерно. Для изготовления раствора подойдет и один миксер, который справится со своими задачами для среднего здания. Выполнять заливку рекомендуем по форме круга – это позволить основанию подниматься постепенно. Последним действием есть выравнивание. Технология этого процесса идентична со стяжкой.
Заполнять фундамент лучше всего по кругу, чтобы весь периметр поднимался постепенно. На финальном этапе бетон выравнивается также как стяжка, чтобы обеспечить более удобную кладку первого ряда кирпича или другого материала. Стоит отметить, что все нормы и требования для расчетов и строительства прописаны в Снип. Так что изучайте документы и только тогда смело приступайте к выполнению работ.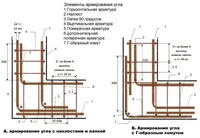
Армирование ленточного фундамента – правила, схемы, инструкции
Возведение фундаментного основания зданий это важнейший этап строительства, который определяет дальнейшую надежность и долговечность постройки. Поэтому при выполнении этой работы не допустима непродуманная экономия на расходах материалов и самовольные изменения проектных решений принятых специалистами.
Ленточные фундаменты пользуются заслуженной популярности при строительстве объектов индивидуальной застройки. Это объясняется возможностью универсального применения для самых различных зданий на большинстве распространенных типов грунтов.
Они отличаются высоким уровнем надежности и возможностью выполнения монтажа своими руками. Ленточные фундаменты нельзя применять для строительства зданий на неустойчивых грунтах, в заболоченной местности и на вечной мерзлоте.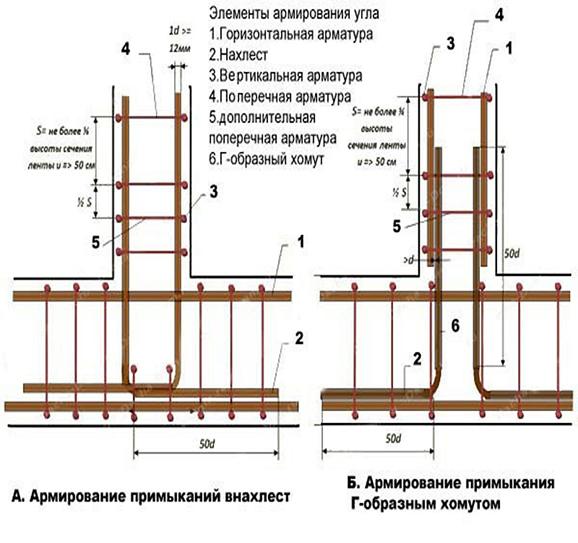
Описание конструкции ленточного фундамента
Несущее основание этого типа представляет собой заглубленную в землю железобетонную монолитную ленту. Она монтируется под все несущие стены и тяжелые перегородки. Глубина заложения фундамента определяется в зависимости от следующих исходных параметров:
- общий вес строительных конструкций здания с учетом снеговых нагрузок, мебели и установленного оборудования;
- тип и строение грунтов на участке;
- глубина залегания грунтовых вод;
- нижняя точка промерзания грунта в холодное время года.
В результате фундамент небольших легких зданий домов быть мелкозаглубленным и иметь нижнюю опору на глубине 500-800 мм. Для тяжелых больших зданий и при наличии подвала подошва монолитной конструкции должна находиться ниже точки промерзания грунта более чем на 400 мм.
Ширина фундаментной ленты в ее верхней части зависит от толщины возводимых стен и должна превышать ее более чем на 100 мм, но в любом случае не мене 300 мм. В нижней части может быть предусмотрено наличие более широкой опорной подошвы, которая устраивается при большом весе строительных конструкций или слабых грунтах. Однако правильный расчет такой опоры довольно сложная инженерная задача. Данные о поперечном сечении фундаментной ленты и об общей массе строительных конструкций позволяют правильно рассчитать конструкцию армирующего каркаса.
В нижней части может быть предусмотрено наличие более широкой опорной подошвы, которая устраивается при большом весе строительных конструкций или слабых грунтах. Однако правильный расчет такой опоры довольно сложная инженерная задача. Данные о поперечном сечении фундаментной ленты и об общей массе строительных конструкций позволяют правильно рассчитать конструкцию армирующего каркаса.
Расчет фундамента должен быть выполнен на профессиональном уровне
Наличие армирующего каркаса повышает прочность фундаментного монолита и позволяет более равномерно распределить весовую нагрузку на грунт. При проектировании элементов здания всегда учитываются реальные данные, на основании которых получают результат способный обеспечить долговечность и надежность постройки.
На основании этого можно сделать вывод, что для разработки проекта необходимы специальные знания и опыт подобных работ. Поэтому выполнение расчетов и определение проектных схем рекомендуется поручить специалисту, а вот монтажные работы можно выполнять самостоятельно. Если только вы не собираетесь построить небольшой сарай, баньку, хозяйственные постройки или легкий гараж.
Если только вы не собираетесь построить небольшой сарай, баньку, хозяйственные постройки или легкий гараж.
Расчет необходимого количества материалов
При определении нужного количества арматуры следует учитывать, что продольные струны и поперечные прутки имеют разный диаметр и цену. Имея проект подсчитать количество необходимого для армирования материала не сложно. Только следует предусмотреть запас 7-10% на остатки в виде коротких обрезков и на нахлесты при соединении прутов на длинных участках.
Если вы производите расчеты самостоятельно, то рекомендуется принять:
- диаметр арматуры 10 мм для продольных участков длиной до 3-х метров;
- 12 мм на участках более 3-х метров;
- поперечная арматура с гладкой поверхностью диаметром 8 мм.
Кроме этого не забудьте приобрести вязальную проволоку (сварка прута для железобетона запрещена), а так же фиксаторы «звездочка» и «опора», которые устанавливаются на каждый крайний прут через каждые 3 метра.
Общее количество продольных армирующих струн определяется по суммарному сечению. Согласно СНиП общая площадь сечения арматуры должна быть не менее 0,1% от поперечного сечения фундаментной ленты. Если в результате вы определите, что для армирования достаточно всего 2-х прутов, то эту количество необходимо увеличить до 4-х. При этом принимая минимальное сечение прутов в 10 мм. Поперечные прутки никаких нагрузок не несут и считаются фиксирующими элементами.
Шаг поперечных прутков (хомутов) должен быть не более трех четвертей высоты фундаментной ленты и меньше 500 мм. В местах примыкания двух прямых конструкций и на углах шаг должен уменьшаться вдвое. Существует много специально разработанных схем вязки углов элементов и примыкающих участков. Перед началом работы рекомендуем с ними ознакомиться.
Что нужно знать про арматуру
Для ленточных фундаментов обычно применяют горячекатаную арматуру классов A-II и A-III с диаметром от 10 мм с периодическим профилем (рифленую), который обеспечивает надежное сцепление металла с бетоном. Пруты класса A-I с гладкой поверхностью и сечением 8-10 мм применяют для изготовления связующих хомутов и перемычек.
Пруты класса A-I с гладкой поверхностью и сечением 8-10 мм применяют для изготовления связующих хомутов и перемычек.
Особенности армирования ленточного фундамента
Для чего нужно армировать ленточный фундамент?
Особенностью мелкозаглубленного облегченного ленточного фундамента является обязательность его армирования. Известно, что бетонные изделия очень прочные на сжатие, менее прочные на сдвиг, и малопрочные на изгиб и разрыв. Компенсируют такие недостатки бетона традиционным способом – созданием композитного материала, в котором одно вещество прекрасно работает на сжатие, а другое – на разрыв. Хорошо сжимаемое вещество дополняют волокнами или стержнями из материала плохо рвущегося и получают новый материал, свойства которого расчетом можно изменять в больших пределах.
Поэтому тонкий слой бетона, известного людям уже более 3 тыс. лет только в XIX веке придумали упрочнить стальной сеткой. Хотя строители знали, что хорошо разрывающаяся глина прекрасно армируется прочной на разрыв соломой.
В случаях, когда на участке неоднородные грунты, армирование ленточного фундамента обеспечит жесткость его рамной конструкции, берущей на себя всю нагрузку от здания и равномерно ее распределяющую.
Общая высота ленточного фундамента обычно от 0,7 – 0,8 м до 1,5 м при ширине от 0,3 до 0,5 м. При длине стены здания от 7 – 10 м такая полоса бетона рассматривается как бетонная балка. Она будет работать на прогиб, когда ее края нагрузить значительно больше, чем середину или наоборот. Т. е. бетон будет нагружен изгибающими усилиями. Защитить балку от разрушения можно поместив в ее толщу в верхней и нижней части продольные стальные или композитные стержни с регулярной профилировкой поверхности. Они за счет профилировки воспримут на себя разрывающие усилия и не дадут растрескаться бетону.
Особенности конструкции армирующего каркаса
Ленточный фундамент фактически состоит из монолитных длинных балок, работающих на изгиб при неравномерных нагрузках сверху от элемен
СП 15. 13330.2012 «Каменные и армокаменные конструкции. Актуализированная редакция СНиП II-22-81»
13330.2012 «Каменные и армокаменные конструкции. Актуализированная редакция СНиП II-22-81»
%PDF-1.5
%
2 0 obj
>
/Metadata 5 0 R
/Outlines 6 0 R
/PageLayout /OneColumn
/StructTreeRoot 7 0 R
/ViewerPreferences >
>>
endobj
5 0 obj
>
stream
2012-02-03T14:33:43+04:002012-02-03T14:33:47+04:00Acrobat PDFMaker 9.1 для Word2012-10-24T16:05:34+04:00uuid:d1a772cf-edd4-4f29-beed-7f00172a1dd1uuid:ae619504-4c71-4ec1-89ee-aac095340796
application/pdf
каменные и армокаменные конструкции, расчетные характеристики материалов, расчетные сопротивления кладки, модули упругости и деформации кладки, упругие характеристики кладки, деформации усадки, коэффициент линейного расширения и трения, расчет элементов конструкций по предельным состояниям первой группы (по несущей способности и второй группы (по образованию и раскрытию трещин, по деформациям)PDF-XChange Viewer [Version: 2. 0 (Build 52.0) (Jun 7 2010; 19:37:04)]D:20120203072304
0 (Build 52.0) (Jun 7 2010; 19:37:04)]D:20120203072304
endstream
endobj
91 0 obj
>
stream
HVM7W]`$0܂Mr(C~H﮷ P [FN0ly2wgЀu?u. j}5iR䭏f{|LcƏ_NWF88:yݫ/yx~8̿gj`cÇc eA=’x
j}5iR䭏f{|LcƏ_NWF88:yݫ/yx~8̿gj`cÇc eA=’x
Frontiers | Генотипирование путем секвенирования Идентификация SNP для сельскохозяйственных культур без эталонного генома: использование картирования на основе транскриптомов в качестве альтернативной стратегии
Введение
За последние два десятилетия технологии секвенирования следующего поколения (NGS) (Mardis, 2008) сделали возможной сборку множества новых эталонных геномов (Ellegren, 2014). Тем не менее, в случае немодельных организмов доступ к разнообразию генома остается проблемой. Секвенирование только части большого генома было предложено как многообещающий способ обойти это ограничение (Narum et al., 2013). Подходы к секвенированию библиотеки с уменьшенным представлением (RRL) позволяют секвенировать часть генома, а также гомологичные области у группы людей. Среди техник RRL сегодня широко используются два основных подхода: подход RAD-seq (Baird et al., 2008; Davey et al., 2011) и подход генотипирования путем секвенирования (GBS) (Elshire et al. , 2011). ), но также доступны несколько других (например, PE-RAD, dd-RAD, 2b-RAD, ezRAD). GBS, как и RAD-seq, снижает сложность генома за счет рестрикционного переваривания, но предлагает упрощенный и более экономичный протокол подготовки библиотеки (Elshire et al., 2011). Эти молекулярные методы были разработаны одновременно с конкретными конвейерами биоинформатики для обработки результирующих исходных последовательностей NGS. Например, конвейер Stacks был разработан в основном для данных RAD-seq (Catchen et al., 2011, 2013), а конвейер TASSEL был разработан для подхода GBS (Glaubitz et al., 2014).
, 2011). ), но также доступны несколько других (например, PE-RAD, dd-RAD, 2b-RAD, ezRAD). GBS, как и RAD-seq, снижает сложность генома за счет рестрикционного переваривания, но предлагает упрощенный и более экономичный протокол подготовки библиотеки (Elshire et al., 2011). Эти молекулярные методы были разработаны одновременно с конкретными конвейерами биоинформатики для обработки результирующих исходных последовательностей NGS. Например, конвейер Stacks был разработан в основном для данных RAD-seq (Catchen et al., 2011, 2013), а конвейер TASSEL был разработан для подхода GBS (Glaubitz et al., 2014).
Следовательно, даже несмотря на то, что данные RAD-seq и GBS могут быть проанализированы с использованием любого из конвейеров, они предпочтительно анализируются с использованием их исходного соответствующего конвейера.Также существует предпочтение каждому подходу РРЛ, которое зависит от заинтересованного «научного сообщества». Например, RAD-seq широко используется для изучения истории эволюции и сохранения диких организмов (Hohenlohe et al.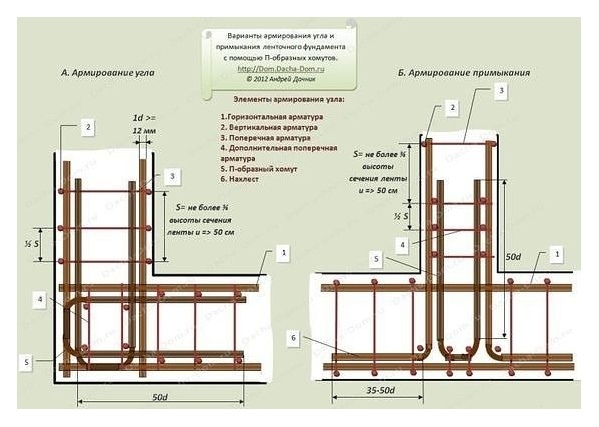 , 2013; Pujolar et al., 2014; Combosch and Vollmer, 2015), тогда как GBS используется исследователями, работающими с культурами и домашние животные. Таким образом, конвейер TASSEL был в первую очередь разработан для выполнения секвенирования с низким охватом гомозиготных образцов (Glaubitz et al., 2014) и для использования в полногеномных ассоциативных исследованиях (Moumouni et al., 2015; Sonah et al., 2015; Упадхьяя и др., 2015). Даже среди сельскохозяйственных культур не все виды являются модельными организмами с эталонным геномом. Когда ссылка недоступна, в Stacks и TASSEL реализуются несколько схожие стратегии для идентификации SNP. Во-первых, идентифицируются и группируются похожие чтения для создания тегов. Во-вторых, создаются сети ТАГов, чтобы определить, какие ТАГ можно рассматривать как альтернативные копии тех же геномных локусов. Эти шаги зависят от нескольких параметров, таких как минимальный охват, чтобы считывание считалось TAG, или от количества несовпадений между двумя TAG, которые следует рассматривать как альтернативные копии одного локуса или разных локусов.
, 2013; Pujolar et al., 2014; Combosch and Vollmer, 2015), тогда как GBS используется исследователями, работающими с культурами и домашние животные. Таким образом, конвейер TASSEL был в первую очередь разработан для выполнения секвенирования с низким охватом гомозиготных образцов (Glaubitz et al., 2014) и для использования в полногеномных ассоциативных исследованиях (Moumouni et al., 2015; Sonah et al., 2015; Упадхьяя и др., 2015). Даже среди сельскохозяйственных культур не все виды являются модельными организмами с эталонным геномом. Когда ссылка недоступна, в Stacks и TASSEL реализуются несколько схожие стратегии для идентификации SNP. Во-первых, идентифицируются и группируются похожие чтения для создания тегов. Во-вторых, создаются сети ТАГов, чтобы определить, какие ТАГ можно рассматривать как альтернативные копии тех же геномных локусов. Эти шаги зависят от нескольких параметров, таких как минимальный охват, чтобы считывание считалось TAG, или от количества несовпадений между двумя TAG, которые следует рассматривать как альтернативные копии одного локуса или разных локусов. Конвейер TASSEL «без эталонного генома» реализован в модуле UNEAK (Universal Network Enabled Analysis Kit) (Lu et al., 2013). SNP идентифицируются путем рисования простых сетей взаимных тегов, которые отличаются несоответствием только на 1 п.н. Значительное влияние параметров конвейера на идентифицированные SNP и выводы популяционной генетики были выделены для Stacks (Catchen et al., 2013; Mastretta-Yanes et al., 2014; Rodríguez-Ezpeleta et al., 2016). Насколько нам известно, влияние метода вызова UNEAK на популяционную генетику еще не исследовано.
Конвейер TASSEL «без эталонного генома» реализован в модуле UNEAK (Universal Network Enabled Analysis Kit) (Lu et al., 2013). SNP идентифицируются путем рисования простых сетей взаимных тегов, которые отличаются несоответствием только на 1 п.н. Значительное влияние параметров конвейера на идентифицированные SNP и выводы популяционной генетики были выделены для Stacks (Catchen et al., 2013; Mastretta-Yanes et al., 2014; Rodríguez-Ezpeleta et al., 2016). Насколько нам известно, влияние метода вызова UNEAK на популяционную генетику еще не исследовано.
Альтернативной стратегией было бы картирование геномных чтений из подходов RRL непосредственно на транскриптоме. Большинство немодельных культур имеют ссылку на транскриптом, которая была создана в первую очередь для исследований транскриптома. Раньше создание транскриптома было сложной задачей (Martin and Wang, 2011; Góngora-Castillo and Buell, 2013), но сегодня доступны новые инструменты, которые позволяют быстро и эффективно получить новую сборку (Grabherr et al. , 2011). Транскриптомы обеспечивают доступ к более длинным последовательностям вокруг SNP, что является очень интересной функцией для дальнейшей проверки SNP и доступа к аннотации геномной области.Таким образом, использование ссылки на транскриптом для считывания карт из подходов RRL (Russell et al., 2013; Combosch and Vollmer, 2015) может быть интересной альтернативой для обнаружения SNP.
, 2011). Транскриптомы обеспечивают доступ к более длинным последовательностям вокруг SNP, что является очень интересной функцией для дальнейшей проверки SNP и доступа к аннотации геномной области.Таким образом, использование ссылки на транскриптом для считывания карт из подходов RRL (Russell et al., 2013; Combosch and Vollmer, 2015) может быть интересной альтернативой для обнаружения SNP.
Однако непросто оценить систематическую ошибку, возникающую в результате использования конвейера вызова SNP, особенно для популяционно-генетических исследований (Hohenlohe et al., 2010; Nielsen et al., 2012; Arnold et al., 2013; Davey et al. , 2013; Gautier et al., 2013; Han et al., 2014; Ilut et al., 2014; Harvey et al., 2015; Rodríguez-Ezpeleta et al., 2016). Поэтому ниже мы сравниваем два набора SNP, полученных из популяций дикого проса с использованием секвенирования GBS. Первый набор SNP был получен через конвейер UNEAK без эталонного генома, а второй набор был получен через конвейер сопоставления с транскриптомом жемчужного проса. Поэтому мы исследовали различия и совпадение SNP, что потребовало оценки структуры популяции и анализа генетического разнообразия.
Поэтому мы исследовали различия и совпадение SNP, что потребовало оценки структуры популяции и анализа генетического разнообразия.
Материалы и методы
Растительный материал
Мы отобрали 48 популяций жемчужного проса [ Pennisetum glaucum (L.) R. Br. ssp. monodii ] из коллекции, хранящейся в IRD (Institut de Recherche pour le Développement, Монпелье, Франция). 48 популяций были выбраны для охвата известного распространения дикого жемчужного проса (рис. 1). Семена выращивали в теплице до цветения, и соцветия 10 растений на популяцию собирали для экстракции ДНК. ДНК экстрагировали с использованием протокола MATAB (модифицированный метод CTAB / β-меркаптоэтанол; Mariac et al., 2006). Набор из 95 ДНК, нормированных до 100 нг / мкл (размер образца на популяцию ≤ 2), был отправлен в Институт геномного разнообразия Корнельского университета для генотипирования GBS.Подробную информацию о протоколе GBS можно найти в другом месте (Elshire et al., 2011; Cronn et al. , 2012). Геномные библиотеки были сконструированы с использованием рестрикционного фермента ApeKI . Полученную библиотеку из 95 сплетений секвенировали с помощью Illumina HiSeq2000. Четыре образца не использовались для последующих анализов из-за большого количества отсутствующих генотипов (> 70%).
, 2012). Геномные библиотеки были сконструированы с использованием рестрикционного фермента ApeKI . Полученную библиотеку из 95 сплетений секвенировали с помощью Illumina HiSeq2000. Четыре образца не использовались для последующих анализов из-за большого количества отсутствующих генотипов (> 70%).
РИСУНОК 1. Географическое распределение 48 популяций дикого жемчужного проса.
Обнаружение SNP и вызов генотипа
UNEAK Трубопровод
необработанных последовательностей были обработаны с помощью модификации конвейера TASSEL-GBS (Glaubitz et al., 2014): трубопровод UNEAK (Lu et al., 2013). При использовании конвейера UNEAK выравнивание тегов с эталонным геномом заменяется созданием пары тегов и сетевой фильтрацией для обнаружения SNP (Lu et al., 2013). Вкратце, хорошие считывания были определены как считывания с точным совпадением штрих-кода без N в 64 битах, следующих за штрих-кодом. Чтения были впоследствии обрезаны до 64 п. н. (без учета штрих-кодов). Уникальные теги TAG с последовательностью длиной 64 п.н., которые присутствовали пять или более раз во всех образцах, были сохранены и использованы для идентификации «пар тегов» с коэффициентом допустимости ошибок (ETR) по умолчанию, равным 0.03, как описано в Lu et al. (2013). Взаимные «ТАГ-пары» с несоответствием всего в 1 п.н. рассматривались как предполагаемые SNP. Оценки правдоподобия для каждого возможного генотипа рассчитывались по формуле 3.8 Etter et al. (2011) и был назначен наиболее вероятный генотип. SNP с частотой минорного аллеля (MAF) ниже 0,05 были исключены. Анализы проводились с помощью TASSEL версии 3.0.157. Окончательный набор SNP (262 928) затем был отфильтрован по глубине покрытия (DP) и проценту отсутствующих данных на SNP (<10%).Мы используем медианное значение охвата по всем SNP в качестве порогового значения для фильтра DP.
н. (без учета штрих-кодов). Уникальные теги TAG с последовательностью длиной 64 п.н., которые присутствовали пять или более раз во всех образцах, были сохранены и использованы для идентификации «пар тегов» с коэффициентом допустимости ошибок (ETR) по умолчанию, равным 0.03, как описано в Lu et al. (2013). Взаимные «ТАГ-пары» с несоответствием всего в 1 п.н. рассматривались как предполагаемые SNP. Оценки правдоподобия для каждого возможного генотипа рассчитывались по формуле 3.8 Etter et al. (2011) и был назначен наиболее вероятный генотип. SNP с частотой минорного аллеля (MAF) ниже 0,05 были исключены. Анализы проводились с помощью TASSEL версии 3.0.157. Окончательный набор SNP (262 928) затем был отфильтрован по глубине покрытия (DP) и проценту отсутствующих данных на SNP (<10%).Мы используем медианное значение охвата по всем SNP в качестве порогового значения для фильтра DP.
Конвейер отображения на основе транскриптомов (TM)
Транскриптом дикого жемчужного проса содержит 50 313 контигов общим размером 36,5 МБ. Этот транскриптом был построен из РНК ранних соцветий, когда дифференциальная экспрессия не была слишком выраженной. Средняя длина контига составляет 725 п.н. ± 732 п.н. (сборка транскриптома).
Этот транскриптом был построен из РНК ранних соцветий, когда дифференциальная экспрессия не была слишком выраженной. Средняя длина контига составляет 725 п.н. ± 732 п.н. (сборка транскриптома).
необработанных последовательностей были сначала обрезаны для получения концов низкого качества (<20), а чтения менее 35 п.н. были удалены с помощью Cutadapt 1.2.1 (Мартин, 2011). Во-вторых, фильтр по среднему качеству чтения был применен с порогом 30. Чтения были сопоставлены с сборкой с помощью BWA версии 0.7.5 (Li and Durbin, 2009) с –n 3, что допускает максимальное количество трех несоответствий. Несопоставленные чтения были удалены с помощью SAMtools версии 0.1.17 (Li et al., 2009). Мы использовали RealignerTargetCreator и IndelRealigner из GATK версии 2.4.7 (DePristo et al., 2011) для обработки отступов. SNP и генотипы вызывали с помощью UnifiedGenotyper. Затем в общей сложности 236 897 SNP были отфильтрованы не более чем на три несоответствия на окно 10 бит / с, был применен фильтр качества отображения (MQ) HARD_TO_VALIDATE [MQ0 ≥ 4 && ((MQ0 / (1. 0 ∗ DP))> 0,1], и фильтрация проводилась для параметров QUAL (качество) и QD (качество по глубине), которые получены из показателей качества Illumina (QUAL ≤ 60; QD ≤ 6,87, квантиль 5%). Затем 121 279 оставшихся SNP были отфильтрованы для DP с использованием среднего значения и процента пропущенных данных на SNP (≤10%). Важно отметить, что дополнительные фильтры качества не могут применяться в конвейере UNEAK, поскольку показатели качества Illumina не используются и не сохраняются в конвейере.Все командные строки доступны в файле дополнительных данных S1, а наборы данных доступны по адресу https://sites.google.com/site/africropproject/data.
0 ∗ DP))> 0,1], и фильтрация проводилась для параметров QUAL (качество) и QD (качество по глубине), которые получены из показателей качества Illumina (QUAL ≤ 60; QD ≤ 6,87, квантиль 5%). Затем 121 279 оставшихся SNP были отфильтрованы для DP с использованием среднего значения и процента пропущенных данных на SNP (≤10%). Важно отметить, что дополнительные фильтры качества не могут применяться в конвейере UNEAK, поскольку показатели качества Illumina не используются и не сохраняются в конвейере.Все командные строки доступны в файле дополнительных данных S1, а наборы данных доступны по адресу https://sites.google.com/site/africropproject/data.
Перекрытие двух наборов данных SNP
Мы выровняли файл Hapmap последовательностей TAG на транскриптоме, используя BWA версии 0.7.5 (Li and Durbin, 2009) с –n 3, что позволило получить максимум три выравнивания. Мы сообщаем только о тегах с уникальным успехом.
Чтобы идентифицировать SNP, общие для двух наборов данных, мы определили TAG среди 21 913 финальных UNEAK SNP, которые совпадают с транскриптомом и извлекают позицию SNP. Затем мы сравнили положение и аллели, чтобы идентифицировать гомологичные SNP в наборе данных TM.
Затем мы сравнили положение и аллели, чтобы идентифицировать гомологичные SNP в наборе данных TM.
Статистика разнообразия и генетическая структура населения
Мы провели большинство анализов в среде R (R Core Team, 2015). Мы выполнили анализ главных компонентов (PCA) с использованием SMARTPCA (Patterson et al., 2006; Price et al., 2006), реализованного в R-пакете SNPRelate (Zheng et al., 2012). Мы использовали R-пакет Adegenet (Jombart and Ahmed, 2011) для оценки значений гетерозиготности и R-пакет Pegas (Paradis, 2010) для F-статистики.Мы использовали программу sNMF для определения структуры популяции (Frichot et al., 2014). Это программное обеспечение дает результаты, аналогичные результатам, полученным с помощью STRUCTURE (Pritchard et al., 2000), но оно намного быстрее и может обрабатывать очень большое количество SNP. Наконец, был рассчитан частотный спектр свернутого участка (SFS), который использовался для оценки w , π и D Tajima (Tajima, 1989).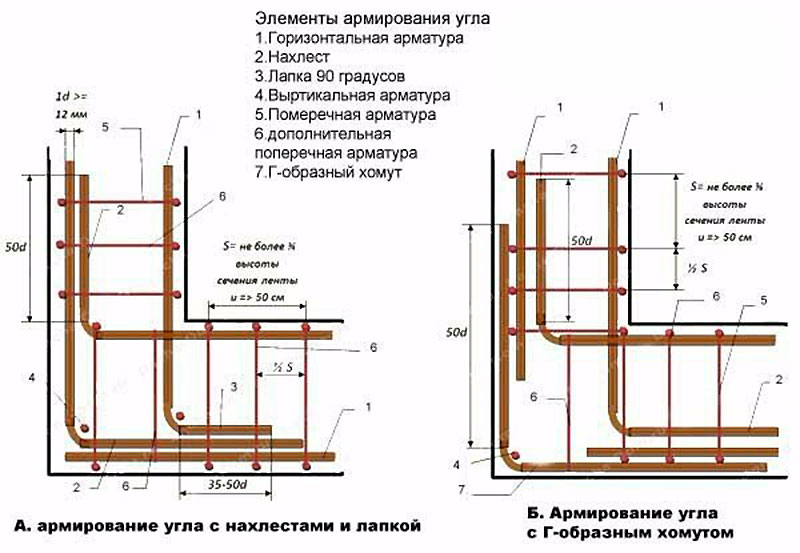 Кроме того, мы оценили SFS, ожидаемую для популяции, находящейся в равновесии в каждом наборе данных (Fu, 1995).
Кроме того, мы оценили SFS, ожидаемую для популяции, находящейся в равновесии в каждом наборе данных (Fu, 1995).
Результаты
Отображение и обнаружение SNP
Оба трубопровода произвели одинаково большое количество SNP. С помощью конвейера UNEAK мы смогли идентифицировать 262 928 двуаллельных SNP. После фильтрации по глубине (DP ≤ 51, фильтрация 50,5%) и отсутствующим данным (NA ≥ 0,1, фильтрация 41,2%) мы получили 21 913 SNP хорошего качества. При использовании подхода TM в общей сложности 16 399 078 очищенных прочтений со средним размером 92 п.н. сопоставлены с 36 918 контигами. Средний охват составил 41,33 ± 44,2, а средний MQ — 24.5. Мы идентифицировали 238 897 двуаллельных SNP со средней глубиной 90 ×, после фильтрации мы получили в общей сложности 22 262 SNP хорошего качества. Специальные фильтры (кластеризация SNP, сопоставление и фильтры качества) из конвейера TM удалили почти 50% SNP, в то время как последующие фильтры для SNP по глубине (DP ≤ 90) и отсутствующих данных (NA ≥ 0,1) удалили 25 и 13,5% соответственно (Рисунок 2 ).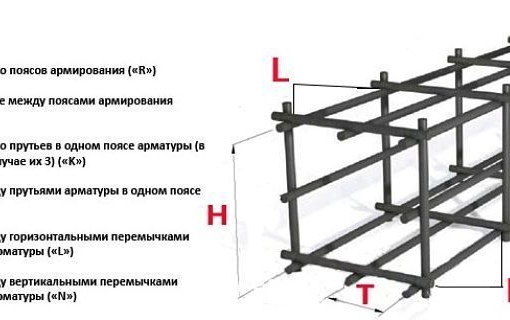
РИСУНОК 2. Доли SNP, удаленные для каждого фильтра, примененного для обоих наборов данных UNEAK и TM.
Окончательные наборы SNP показали, что качество этих двух подходов было эквивалентным. Окончательный набор UNEAK из 21 913 SNP имел среднее значение DP на сайт и на образец, равное 7,24 ± 3,63 sd, и среднюю частоту пропусков на образец 0,04 ± 0,04 sd. Окончательный набор TM из 22 262 SNP имел среднее значение DP на сайт и на образец 8,68 ± 12 sd и среднюю частоту пропусков на образец 0,03 ± 0,03 sd. В финальном наборе TM 56% SNP были обнаружены на расстоянии 64 п.н. Коэффициенты пропусков на выборку между наборами данных UNEAK и TM сильно коррелировали ( r = 0.95). Тем не менее, у нас было в среднем 70% завышенного числа недостающих данных с UNEAK, так как среднее соотношение недостающих данных UNEAK: TM составляло 1,7 ± 1,8 sd.
Кроме того, мы протестировали прямое картирование 262 928 UNEAK TAG 64 п.н. на транскриптоме. Всего 21 410 локусов TAG (8%) картированы на 13 177 контигах транскриптомов (26%). Сопоставление было относительно хорошим, поскольку 94% сопоставленных тегов имели уникальное совпадение, из которых 96% имели идеальное совпадение в 64 п.н. Средний MQ этих уникальных совпадений составил 34 ± 9 SD. Среди 21 943 тегов хорошего качества мы обнаружили 3146 тегов (14%), которые хорошо согласовывались с 2382 (5%) контигами.Среди них мы получили 822 SNP, общих для двух наборов данных. Почти все UNEAK SNP имели MAF> 0,05 (дополнительный рисунок S1 и таблица S1). Коэффициент корреляции между частотами аллелей, оцененными обоими конвейерами для общих SNP, был очень высоким ( r = 0,98).
Всего 21 410 локусов TAG (8%) картированы на 13 177 контигах транскриптомов (26%). Сопоставление было относительно хорошим, поскольку 94% сопоставленных тегов имели уникальное совпадение, из которых 96% имели идеальное совпадение в 64 п.н. Средний MQ этих уникальных совпадений составил 34 ± 9 SD. Среди 21 943 тегов хорошего качества мы обнаружили 3146 тегов (14%), которые хорошо согласовывались с 2382 (5%) контигами.Среди них мы получили 822 SNP, общих для двух наборов данных. Почти все UNEAK SNP имели MAF> 0,05 (дополнительный рисунок S1 и таблица S1). Коэффициент корреляции между частотами аллелей, оцененными обоими конвейерами для общих SNP, был очень высоким ( r = 0,98).
Генетическая структура и генетическое разнообразие
Два набора данных показали очень похожие выводы о генетической структуре. Мы определили K = 3 группы населения географически в западном, центральном и восточном кластерах с обоими наборами данных (рис. 3).Корреляция между значениями примеси из обоих подходов в пределах каждого кластера была высокой: r > 0,99. Результаты PCA были аналогичными (рис. 3). Оба набора данных показали одни и те же три географических кластера, и корреляция между координатами PCA была очень высокой ( r > 0,99). При сравнении UNEAK и TM PCA только один образец (образец 5726B1) находился в другом положении на двух графиках. У этого человека было в 17 раз больше пропущенных данных с набором данных UNEAK, чем с набором данных TM, несмотря на то, что количество пропущенных данных <0.05%. Это очень высокое соотношение отсутствующих данных между наборами данных может объяснить его статус выброса. В более общем плане регрессия координат PCA между двумя конвейерами показала, что большинство людей, квалифицированных как незначительные выбросы, имели в три раза больше отсутствующих данных в конвейере UNEAK, чем в конвейере TM. Однако в целом мы наблюдали очень хорошее индивидуальное качество и очень сильный конгруэнтный вывод о структуре населения независимо от того, какой трубопровод использовался.
Результаты PCA были аналогичными (рис. 3). Оба набора данных показали одни и те же три географических кластера, и корреляция между координатами PCA была очень высокой ( r > 0,99). При сравнении UNEAK и TM PCA только один образец (образец 5726B1) находился в другом положении на двух графиках. У этого человека было в 17 раз больше пропущенных данных с набором данных UNEAK, чем с набором данных TM, несмотря на то, что количество пропущенных данных <0.05%. Это очень высокое соотношение отсутствующих данных между наборами данных может объяснить его статус выброса. В более общем плане регрессия координат PCA между двумя конвейерами показала, что большинство людей, квалифицированных как незначительные выбросы, имели в три раза больше отсутствующих данных в конвейере UNEAK, чем в конвейере TM. Однако в целом мы наблюдали очень хорошее индивидуальное качество и очень сильный конгруэнтный вывод о структуре населения независимо от того, какой трубопровод использовался.
РИСУНОК 3. Выводы структуры населения для обоих наборов данных SNP UNEAK (слева) и TM (справа) с использованием программного обеспечения sNMF (A, B) и PCA (C, D). Западный кластер выделен красным, Центральный — синим, а Восточный — зеленым.
Выводы структуры населения для обоих наборов данных SNP UNEAK (слева) и TM (справа) с использованием программного обеспечения sNMF (A, B) и PCA (C, D). Западный кластер выделен красным, Центральный — синим, а Восточный — зеленым.
Напротив, оценка генетического разнообразия затрагивалась по-разному в зависимости от конвейера. Значения гетерозиготности были почти в два раза выше с набором данных UNEAK, чем с набором данных TM (Таблица 1). Для F-статистики F IS был немного, но значительно выше с набором данных TM, а F ST был значительно (в два раза) ниже.Когда мы сравнили наблюдаемую SFS и ожидаемую SFS для популяции в равновесии, набор данных UNEAK явно не извлек ожидаемое количество низкочастотных SNP (рис. 4). С другой стороны, TM SFS, похоже, переоценила их количество. В результате π было в 2,2 раза выше с набором данных UNEAK, а значения Tajima D , следовательно, значительно различались с положительным значением D Tajima, равным 2,74 для UNEAK и отрицательным значением -0,65 для набора данных TM.
РИСУНОК 4. Частотный спектр свернутых участков для обоих наборов данных SNP: (A) TM (красным) и его ожидаемая нейтральная SFS черным цветом; (B) UNEAK (синим) и его ожидаемая нейтральная SFS черным.
ТАБЛИЦА 1. Сводка статистики разнообразия для двух наборов данных SNP.
Обсуждение
В этом исследовании мы сравнили два конвейера биоинформатики и их влияние на статистику популяционной генетики. Изучение геномного разнообразия все еще является сложной задачей для немодельных организмов с большими геномами.Подходы секвенирования RRL, такие как подходы RNA-seq и GBS, были предложены для уменьшения сложности генома. Полученные данные NGS могут обрабатываться различными конвейерами, включая Stacks и TASSEL. Здесь мы предпочтительно использовали конвейер TASSEL, потому что он наиболее часто используется для изучения сельскохозяйственных культур. Поэтому мы сначала использовали подход UNEAK, реализованный в TASSEL, и предложили и протестировали альтернативную стратегию, в которой геномные чтения NGS были непосредственно отображены на транскриптоме жемчужного проса.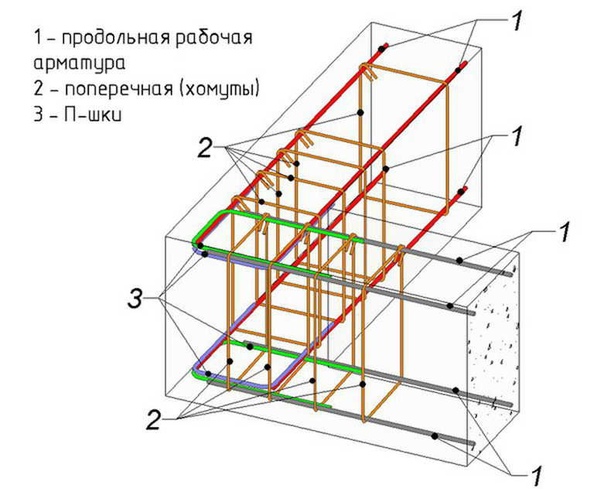 Эта стратегия основывалась на наблюдении, что транскриптомы видов становятся все более доступными благодаря исследованиям транскрипции и что было бы выгодно их использовать (Russell et al., 2013; Combosch and Vollmer, 2015). Это позволяет избежать использования сборки ДНК de novo и имеет преимущество использования эталонного генома, например, для доступа к более длинной последовательности вокруг сайтов SNP, и имеет большую вероятность обнаружения мишеней для отбора (Hancock et al., 2011).
Эта стратегия основывалась на наблюдении, что транскриптомы видов становятся все более доступными благодаря исследованиям транскрипции и что было бы выгодно их использовать (Russell et al., 2013; Combosch and Vollmer, 2015). Это позволяет избежать использования сборки ДНК de novo и имеет преимущество использования эталонного генома, например, для доступа к более длинной последовательности вокруг сайтов SNP, и имеет большую вероятность обнаружения мишеней для отбора (Hancock et al., 2011).
GBS читает предвзятое отношение к регионам кодирования
Качество наших двух финальных наборов данных такое же хорошее, как и у наборов данных, используемых в других популяционно-генетических исследованиях, с окончательным охватом от 5 до 10 и коэффициентом пропущенных значений ниже 0,3. Многие наборы данных RRL могут иметь низкий охват в исследованиях, дизайн которых нацелен на большее количество людей или локусов, чтобы повысить точность популяционных генетических параметров (Alex Buerkle and Gompert, 2013).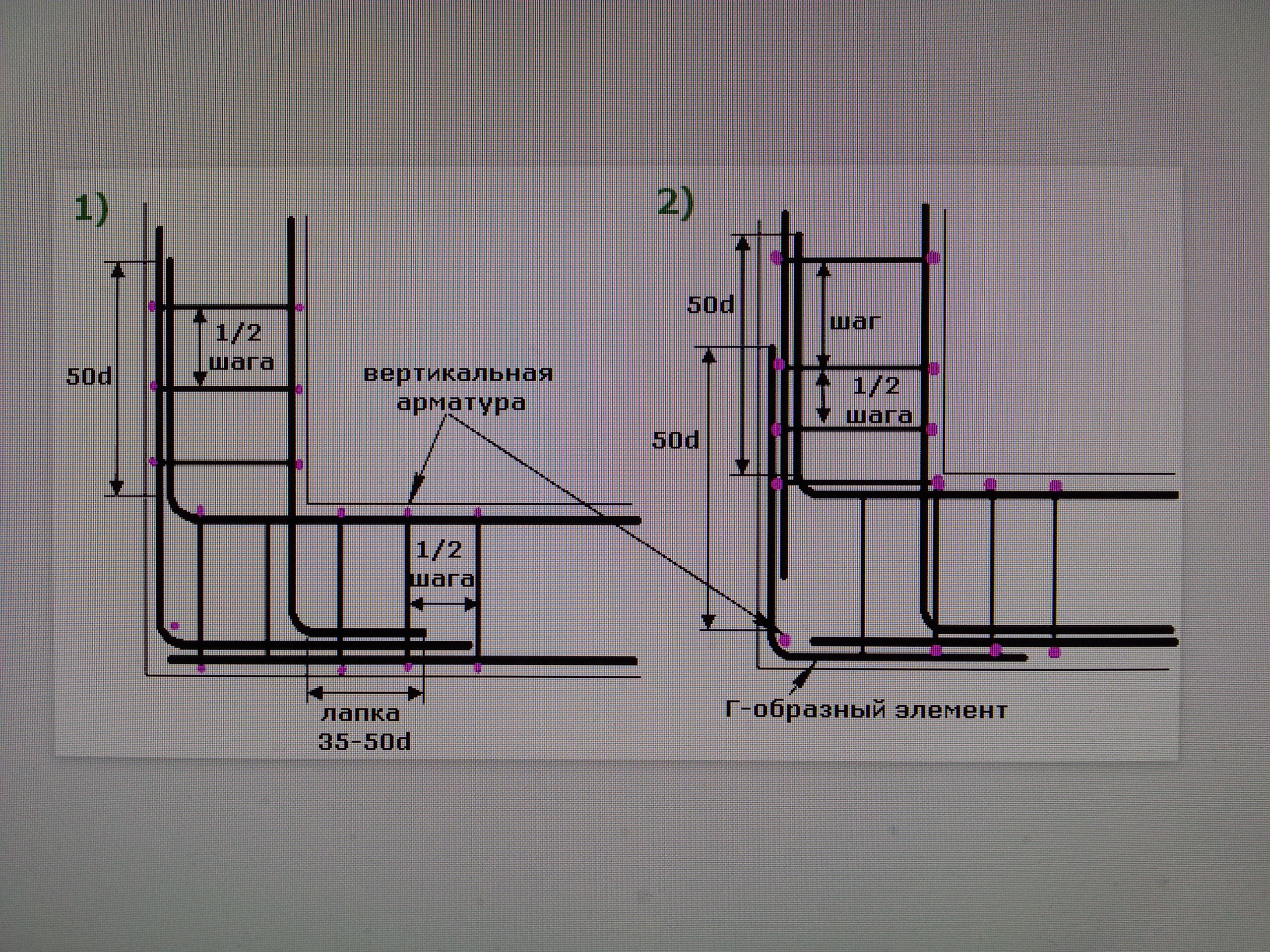
Неожиданно мы обнаружили, что незначительное количество UNEAK TAG отображается в транскриптоме.Combosch и Vollmer (2015) обнаружили, что около 15% локусов RAD картированы на 10% контигов транскриптомов. Наши результаты аналогичны для 8% локусов TAG, которые картированы на 26% контигов транскриптомов. Первоначально мы ожидали очень низкой скорости картирования, поскольку мы картировали только экспрессированный геном. Одним из возможных объяснений является выбор используемого рестрикционного фермента. В нашем и многих других исследованиях использовался фермент ApeKI с подходом GBS (Elshire et al., 2011; Lu et al., 2013). Его чувствительность к метилированию позволила исключить из эксперимента повторяющиеся метилированные участки геномики (Sonah et al., 2013). У эукариот неметилированные сайты преимущественно обнаруживаются в кодирующих областях (Phillips, 2008). В популяциях Populus 27% сайтов рестрикции из всего генома были восстановлены с использованием ApeKI для GBS, из которых 70% попали в аннотированные гены (Schilling et al. , 2014). У черешни ( Prunus avium L.) 66% SNP были обнаружены в генных регионах (Guajardo et al., 2015). В настоящем исследовании на основе генома жемчужного проса, оцененного в 1,8 G (Xin Liu, BGI, личное сообщение), и размера эталонного транскриптома в 36.5 МБ, мы опросили только 2% генома. Мы обнаружили, что 6–7% считываний на образец, сопоставленных с эталоном транскриптома, и 8% локусов UNEAK TAG также были выровнены, что в три-четыре раза больше, чем ожидаемые 2%. Эти результаты согласуются с сообщениями о смещении фермента ApeKI в сторону кодирующих областей в предыдущих исследованиях (Schilling et al., 2014; Guajardo et al., 2015).
, 2014). У черешни ( Prunus avium L.) 66% SNP были обнаружены в генных регионах (Guajardo et al., 2015). В настоящем исследовании на основе генома жемчужного проса, оцененного в 1,8 G (Xin Liu, BGI, личное сообщение), и размера эталонного транскриптома в 36.5 МБ, мы опросили только 2% генома. Мы обнаружили, что 6–7% считываний на образец, сопоставленных с эталоном транскриптома, и 8% локусов UNEAK TAG также были выровнены, что в три-четыре раза больше, чем ожидаемые 2%. Эти результаты согласуются с сообщениями о смещении фермента ApeKI в сторону кодирующих областей в предыдущих исследованиях (Schilling et al., 2014; Guajardo et al., 2015).
Выявлено влияние трубопроводов на SNP
Специфика трубопровода влияет на количество обнаруженных SNP и их характеристики распределения.Существуют серьезные различия в том, как вызываются SNP между конвейерами, потому что конвейеры несколько по-разному работают с ошибками секвенирования, значениями базового качества, вызовом SNP и методами вызова генотипа и, в нашем случае, построением каталога TAG по сравнению с отображением транскриптома. Теперь мы рассмотрим некоторые различия между двумя подходами и то, как такие различия могут повлиять на наши результаты.
Теперь мы рассмотрим некоторые различия между двумя подходами и то, как такие различия могут повлиять на наши результаты.
Среди всех параметров, которые могут повлиять на обнаружение SNP, покрытие является одним из наиболее важных. Например, ожидается, что частота ошибок увеличится при низком охвате (<20 ×; Andrews and Luikart, 2014).Чтобы ограничить влияние покрытия в обоих наших конвейерах, мы отфильтровали SNP с глубиной выше среднего значения для каждого набора данных (51 × для UNEAK, 90 × для TM). Оба окончательных набора данных имели одинаковый охват и одинаковую частоту пропусков. Таким образом, в этом смысле это мало повлияет на количество обнаруженных SNP и оценки популяционной генетики между наборами данных.
Еще одно возможное смещение происходит из-за повторяющихся участков генома, таких как паралоги, и его не всегда легко идентифицировать с помощью данных NGS. Для уменьшения эффекта неопознанных паралогов можно использовать различные фильтры.Ожидается, что паралоговые области будут соответствовать множеству мест в геноме (Hohenlohe et al. , 2012), и ожидается, что SNP внутри генов паралогов будут демонстрировать более двух аллелей (Freedman et al., 2014). Мы рассматривали только двуаллельные локусы в двух наборах данных, поскольку в подходах RRL проблема паралогов может быть эффективно решена с помощью фильтрации на основе плоидности (Ilut et al., 2014). Благодаря подходу TM мы смогли применить дополнительный фильтр к MQ, чтобы уменьшить количество паралоговых областей. Однако при сопоставлении UNEAK TAGs с транскриптомом мы обнаружили, что 94% TAG, которые имели попадание, отображаются в уникальную позицию.Это говорит о том, что даже если фильтр отображения не может быть применен, вероятность вызова паралогов с конвейером UNEAK относительно низка, и фильтрация на основе плоидности, таким образом, кажется достаточной, чтобы избежать смещения паралога.
, 2012), и ожидается, что SNP внутри генов паралогов будут демонстрировать более двух аллелей (Freedman et al., 2014). Мы рассматривали только двуаллельные локусы в двух наборах данных, поскольку в подходах RRL проблема паралогов может быть эффективно решена с помощью фильтрации на основе плоидности (Ilut et al., 2014). Благодаря подходу TM мы смогли применить дополнительный фильтр к MQ, чтобы уменьшить количество паралоговых областей. Однако при сопоставлении UNEAK TAGs с транскриптомом мы обнаружили, что 94% TAG, которые имели попадание, отображаются в уникальную позицию.Это говорит о том, что даже если фильтр отображения не может быть применен, вероятность вызова паралогов с конвейером UNEAK относительно низка, и фильтрация на основе плоидности, таким образом, кажется достаточной, чтобы избежать смещения паралога.
Статистическая обработка последовательностей NGS для данного генотипа основана на предполагаемом независимом отрисовке неизбыточных считываний одного гена. Несколько артефактов могут смещать вероятность генотипа, потому что считывания не ведут себя как основная статистическая гипотеза: одно считывание может быть дубликатом (не независимым), альтернативный аллель может отсутствовать (неслучайный рисунок) или отображение двух разных, но похожих генов (ни одного гена) по одной ссылке. Ни один из конвейеров не очень легко справляется с возникновением статистической независимости чтения. Конвейеры, разработанные для подходов RRL, не смогли справиться с выпадением аллелей и ошибочно принять гетерозиготное присутствие / отсутствие за гомозиготное присутствие / отсутствие (Davey et al., 2013). Совсем недавно был разработан конвейер для работы с доминантными и кодоминантными маркерами (Fu et al., 2013). Тем не менее, с обоими нашими подходами доминантный маркер (т. Е. Мутация в сайте рестрикции, приводящая к выпадению аллеля) привел бы к вызову гомозиготы.Повторяющиеся чтения происходят, когда во время подготовки банка ДНК два чтения происходят из одной ДНК путем дупликации ПЦР. Дубликаты ПЦР по определению считываются, начиная с одной и той же позиции отображения. Влияние дубликатов ПЦР на оценку популяционной генетики уже обсуждалось (Arnold et al., 2013; Davey et al., 2013; Gautier et al., 2013). По конструкции в RRL, основанном на рестрикционных ферментах, считывания будут начинаться с той же позиции картирования, которая является сайтом RE, поэтому применение фильтра дубликатов ПЦР будет невозможно, если не будет использован подход парного секвенирования и случайного разделения (Davey et al.
Ни один из конвейеров не очень легко справляется с возникновением статистической независимости чтения. Конвейеры, разработанные для подходов RRL, не смогли справиться с выпадением аллелей и ошибочно принять гетерозиготное присутствие / отсутствие за гомозиготное присутствие / отсутствие (Davey et al., 2013). Совсем недавно был разработан конвейер для работы с доминантными и кодоминантными маркерами (Fu et al., 2013). Тем не менее, с обоими нашими подходами доминантный маркер (т. Е. Мутация в сайте рестрикции, приводящая к выпадению аллеля) привел бы к вызову гомозиготы.Повторяющиеся чтения происходят, когда во время подготовки банка ДНК два чтения происходят из одной ДНК путем дупликации ПЦР. Дубликаты ПЦР по определению считываются, начиная с одной и той же позиции отображения. Влияние дубликатов ПЦР на оценку популяционной генетики уже обсуждалось (Arnold et al., 2013; Davey et al., 2013; Gautier et al., 2013). По конструкции в RRL, основанном на рестрикционных ферментах, считывания будут начинаться с той же позиции картирования, которая является сайтом RE, поэтому применение фильтра дубликатов ПЦР будет невозможно, если не будет использован подход парного секвенирования и случайного разделения (Davey et al. , 2013), но недавно был предложен новый протокол, в котором были введены «теги адаптера», позволяющие распознавать дубликаты ПЦР (Tin et al., 2015). В заключение, для обоих подходов, которые мы использовали, фильтрация дубликатов ПЦР была невозможна, и поэтому мы ожидали, что наборы данных UNEAK и TM недооценивают гетерозиготность. Это согласуется с сильной корреляцией, наблюдаемой между оценками частот обоими подходами для общих SNP.
, 2013), но недавно был предложен новый протокол, в котором были введены «теги адаптера», позволяющие распознавать дубликаты ПЦР (Tin et al., 2015). В заключение, для обоих подходов, которые мы использовали, фильтрация дубликатов ПЦР была невозможна, и поэтому мы ожидали, что наборы данных UNEAK и TM недооценивают гетерозиготность. Это согласуется с сильной корреляцией, наблюдаемой между оценками частот обоими подходами для общих SNP.
Количество SNP, разрешенных в пределах окна генома, важно, поскольку области со слишком большим количеством SNP не являются надежными и могут (i) содержать много ошибок секвенирования, (ii) быть связаны с паралогами.Внутри конвейера TM мы применили фильтр кластеризации SNP с не более чем тремя SNP на 10 бит. Тем не менее, он позволял читать большое количество SNP при чтении 100 б.п. Например, в наборах данных TM 56% SNP находились на расстоянии менее 64 пбайт. Поскольку подход UNEAK допускает только 1 SNP на 64 pb, более 50% SNP TM будут автоматически отброшены конвейером UNEAK.
Однако эти два конвейера сильно различаются по скорости вызова редких вариантов. Даже если базовое качество выше 30 с платформой секвенирования ILLUMINA, т.е.е., одна ошибка на каждые 103 базиса, с объемом сгенерированных данных, это привело к появлению множества ошибок. Вызов редких вариантов (или нет) будет зависеть от алгоритма вызова SNP и генотипа, реализованного в программном обеспечении, а также от того, как учитывается частота ошибок секвенирования (Han et al., 2014). Для некоторых конвейеров оценка частоты ошибок считается постоянной по всему геному, в то время как другие конвейеры оценивают частоту ошибок для каждой базы (Hohenlohe et al., 2011). Оценки частоты ошибок также могут учитывать зависимость между ошибками секвенирования (или нет; Han et al., 2014). В программном обеспечении GATK предполагается, что ошибки секвенирования не зависят друг от друга и учитываются охват и базовое качество. Таким образом, если покрытие не составляет примерно 10 × на сайт на образец, GATK с UnifiedGenotyper может недооценивать редкие варианты (Han et al. , 2014), тогда как UNEAK обрабатывает ошибки секвенирования иначе. Чтобы решить эту проблему, конвейер UNEAK использует минимальную ETR в 3% для вызова вариантов. Этот ETR оказывает прямое влияние на истинно низкочастотные варианты: с ошибочным SNP истинный SNP отбрасывается.Такой способ обработки частоты ошибок секвенирования может быть основной причиной, по которой UNEAK SFS недооценивает низкочастотные SNP по сравнению с ожидаемым распределением с популяцией в равновесии. Это также объяснило бы, почему используется так мало общих SNP, поскольку в обоих наборах данных можно найти только частые SNP, что подтверждено распределением MAF, наблюдаемым для общих SNP.
, 2014), тогда как UNEAK обрабатывает ошибки секвенирования иначе. Чтобы решить эту проблему, конвейер UNEAK использует минимальную ETR в 3% для вызова вариантов. Этот ETR оказывает прямое влияние на истинно низкочастотные варианты: с ошибочным SNP истинный SNP отбрасывается.Такой способ обработки частоты ошибок секвенирования может быть основной причиной, по которой UNEAK SFS недооценивает низкочастотные SNP по сравнению с ожидаемым распределением с популяцией в равновесии. Это также объяснило бы, почему используется так мало общих SNP, поскольку в обоих наборах данных можно найти только частые SNP, что подтверждено распределением MAF, наблюдаемым для общих SNP.
Таким образом, мы выделили две основные причины небольшого количества общих SNP: (i) ограничение не более чем одного SNP в пределах 64 пар оснований, и (ii) обнаружение редких вариантов UNEAK, которые представляют большинство полиморфизм, ожидаемый для популяции в состоянии равновесия.В итоге мы получили относительно немного общих SNP.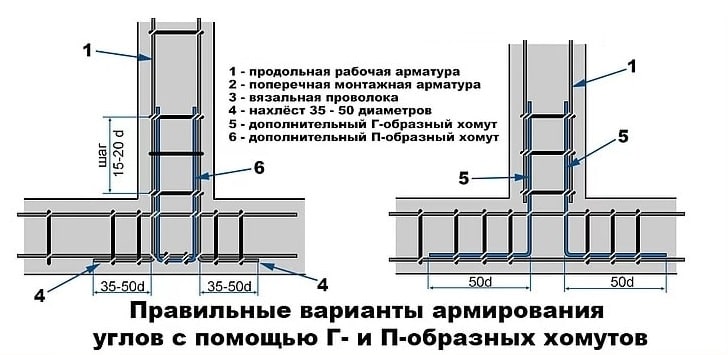 Однако корреляция частот аллелей между этими SNP была очень высокой.
Однако корреляция частот аллелей между этими SNP была очень высокой.
Влияние трубопроводов на оценки разнообразия
Другой важный вопрос — как определенные характеристики идентифицированных нами SNP повлияют на оценки популяционной генетики. Появляется все больше литературы о том, как такие параметры, как количество несовпадений, разрешенных для сборки считываний в ортологичных локусах с подходами RRL, будут влиять на количество идентифицированных SNP и результаты популяции.Большинство доступных исследований сосредоточено на влиянии параметров трубопровода Stacks (Catchen et al., 2013; Mastretta-Yanes et al., 2014). Например, допуск небольшого количества несовпадений приведет к созданию большего количества локусов, чем в реальной жизни, и, наоборот, разрешение слишком большого количества несовпадений приведет к слиянию паралогов. Слишком строгие требования могут увеличить количество ошибок при генотипировании (Mastretta-Yanes et al., 2014) и переоценить гомозиготность (Ilut et al. , 2014). Это также могло повлиять на определение структуры населения (Harvey et al., 2015; Родригес-Эзпелета и др., 2016). Поскольку конвейер UNEAK допускает несоответствие только 1 б.п., это максимальный уровень строгости для конвейера RRL. Тем не менее, мы не заметили никакого влияния на структуру населения, и мы наблюдали очень высокую конгруэнтность в структуре населения в двух наборах данных и с двумя разными методами: байесовским методом и PCA. Эти результаты аналогичны результатам, полученным Rodríguez-Ezpeleta et al. (2016).
, 2014). Это также могло повлиять на определение структуры населения (Harvey et al., 2015; Родригес-Эзпелета и др., 2016). Поскольку конвейер UNEAK допускает несоответствие только 1 б.п., это максимальный уровень строгости для конвейера RRL. Тем не менее, мы не заметили никакого влияния на структуру населения, и мы наблюдали очень высокую конгруэнтность в структуре населения в двух наборах данных и с двумя разными методами: байесовским методом и PCA. Эти результаты аналогичны результатам, полученным Rodríguez-Ezpeleta et al. (2016).
Основное различие между трубопроводами, которое мы наблюдали, касалось выявления низкочастотных вариантов.Мы обнаружили, что конвейер UNEAK не может восстанавливать редкие варианты, в то время как шаблон SFS для частых вариантов был одинаковым для разных конвейеров. Таким образом, методы, основанные на «более частых аллелях», такие как подходы к популяционной структуре, привели к аналогичным результатам. С другой стороны, некоторые статистические данные, использующие низкочастотные варианты, значительно различались в зависимости от используемого набора данных. Тест Tajima D (Tajima, 1989) основан на паттерне SFS, где избыток редких вариантов является признаком расширения популяции или положительного отбора, и, наоборот, сокращение редких вариантов является признаком сокращения или балансирования популяции. выбор.Оба конвейера дали резко контрастирующие результаты, варьирующиеся от общей отрицательной сигнатуры значения -0,65 до положительной сигнатуры значения 2,74. Беспристрастный SFS имеет решающее значение для популяционной генетики. Методы, используемые для изучения истории популяции, включая узкие места или события роста, основаны на разнице между аллельным разнообразием и гетерозиготностью и, следовательно, зависят от идентификации редких вариантов. Более того, SFS широко используются для проверки сигнатуры выбора с использованием Tajima D , но также и другие тесты, такие как тест CLR (Nielsen et al., 2009). С такими тестами, основанными на SFS, вызывающие конвейеры могут значительно повлиять на участки генома, которые оказались под отбором.
Тест Tajima D (Tajima, 1989) основан на паттерне SFS, где избыток редких вариантов является признаком расширения популяции или положительного отбора, и, наоборот, сокращение редких вариантов является признаком сокращения или балансирования популяции. выбор.Оба конвейера дали резко контрастирующие результаты, варьирующиеся от общей отрицательной сигнатуры значения -0,65 до положительной сигнатуры значения 2,74. Беспристрастный SFS имеет решающее значение для популяционной генетики. Методы, используемые для изучения истории популяции, включая узкие места или события роста, основаны на разнице между аллельным разнообразием и гетерозиготностью и, следовательно, зависят от идентификации редких вариантов. Более того, SFS широко используются для проверки сигнатуры выбора с использованием Tajima D , но также и другие тесты, такие как тест CLR (Nielsen et al., 2009). С такими тестами, основанными на SFS, вызывающие конвейеры могут значительно повлиять на участки генома, которые оказались под отбором.
Различия в количестве обнаруженных редких вариантов также влияют на F-статистику в дополнение к гетерозиготности. F ST зависит от частоты аллеля, низкая F ST ожидается для низкочастотных вариантов. Следовательно, интеграция более редких вариантов приводит к добавлению низкого значения F ST и, таким образом, к снижению среднего значения F ST .Первым и наиболее простым следствием будет затруднение сравнения оценок разнообразия, полученных с помощью различных трубопроводов, что является важной проблемой в сравнительных исследованиях (Ilut et al., 2014; Harvey et al., 2015). Еще один очень «модный» подход со времен NGS — это метод обнаружения выбросов F ST для обнаружения генов при отборе. Был разработан ряд тестов с выбросами F ST , которые широко используются для обнаружения генов-кандидатов (Beaumont and Nichols, 1996; Vitalis et al., 2001; Бомонт и Болдинг, 2004 г . ; Фолль и Гаджотти, 2008; Bonhomme et al., 2010; Гюнтер и Куп, 2013; Duforet-Frebourg et al., 2014) и основаны на ожидаемом распределении F ST . Недооценка редких вариантов повлияет на общее распределение F ST и, следовательно, может повлиять на эти критерии отбора.
; Фолль и Гаджотти, 2008; Bonhomme et al., 2010; Гюнтер и Куп, 2013; Duforet-Frebourg et al., 2014) и основаны на ожидаемом распределении F ST . Недооценка редких вариантов повлияет на общее распределение F ST и, следовательно, может повлиять на эти критерии отбора.
Несомненно, что некоторые различия в результатах оценок разнообразия, наблюдаемые между наборами данных, связаны с тем, что конвейер UNEAK опрашивает кодирующие и некодирующие области, тогда как конвейер TM опрашивает только кодирующие области.Однако, как и другие авторы, мы ранее наблюдали, что использование APEKI смещает обнаружение SNP в сторону кодирующих областей (Combosch and Vollmer, 2015; Guajardo et al., 2015). В целом, мы полагаем, что систематическая ошибка в оценках разнообразия в основном является результатом свойств трубопроводов. Предвзятые SFS являются результатом различных параметров, включая формулу оценки частоты ошибок и строгость, разрешенную для слияния TAG. Все больше исследований показывают, что определение генотипа может больше не требоваться для данных NGS (Nielsen et al., 2011; Фумагалли и др., 2013; Han et al., 2014). Nielsen et al. (2011) указали, что до сих пор не существует удовлетворительного алгоритма определения генотипа, который привел бы к беспристрастной SFS. Эти авторы предложили прямой подход, реализованный в программном обеспечении ANGSD (Korneliussen et al., 2014), который не предназначен для вызова генотипов, и этот подход был расширен модифицированным PCA (Fumagalli et al., 2013) и подходом оценки примесей на основе генотипа. правдоподобия (Skotte et al., 2013). Однако это программное обеспечение работает только с файлами BAM в качестве входных и справочных.Учитывая это ограничение, нельзя использовать SNP, полученные из конвейера UNEAK, тогда как наш конвейер TM может интегрировать такой анализ.
Все больше исследований показывают, что определение генотипа может больше не требоваться для данных NGS (Nielsen et al., 2011; Фумагалли и др., 2013; Han et al., 2014). Nielsen et al. (2011) указали, что до сих пор не существует удовлетворительного алгоритма определения генотипа, который привел бы к беспристрастной SFS. Эти авторы предложили прямой подход, реализованный в программном обеспечении ANGSD (Korneliussen et al., 2014), который не предназначен для вызова генотипов, и этот подход был расширен модифицированным PCA (Fumagalli et al., 2013) и подходом оценки примесей на основе генотипа. правдоподобия (Skotte et al., 2013). Однако это программное обеспечение работает только с файлами BAM в качестве входных и справочных.Учитывая это ограничение, нельзя использовать SNP, полученные из конвейера UNEAK, тогда как наш конвейер TM может интегрировать такой анализ.
Заключение
Мы продемонстрировали возможности и обсудили преимущества и недостатки двух конвейеров, используемых для обнаружения SNP, когда ссылка на геном недоступна. Мы обнаружили, что конвейер UNEAK с небольшой и простой работой по биоинформатике может эффективно идентифицировать большое количество SNP, а также выделять генетическую кластеризацию.Однако мы наблюдали заметную недооценку редких вариантов, что могло повлиять на оценку популяционной генетики и обнаружение отбора. Поэтому мы призываем исследователей уделять больше внимания SFS. Ссылка на отображение транскриптома была менее предвзятой в этом смысле, и, что более важно, такую стратегию можно было использовать в сочетании с текущими подходами без вызова генотипа для дальнейшего снижения систематической ошибки SFS. Альтернативная стратегия имеет дополнительное преимущество, позволяющее получить доступ к последовательностям, окружающим SNP, для дальнейшего геномного исследования.Более того, поскольку используется несколько SNP, оба набора данных могут быть объединены, что значительно увеличивает количество используемых SNP.
Мы обнаружили, что конвейер UNEAK с небольшой и простой работой по биоинформатике может эффективно идентифицировать большое количество SNP, а также выделять генетическую кластеризацию.Однако мы наблюдали заметную недооценку редких вариантов, что могло повлиять на оценку популяционной генетики и обнаружение отбора. Поэтому мы призываем исследователей уделять больше внимания SFS. Ссылка на отображение транскриптома была менее предвзятой в этом смысле, и, что более важно, такую стратегию можно было использовать в сочетании с текущими подходами без вызова генотипа для дальнейшего снижения систематической ошибки SFS. Альтернативная стратегия имеет дополнительное преимущество, позволяющее получить доступ к последовательностям, окружающим SNP, для дальнейшего геномного исследования.Более того, поскольку используется несколько SNP, оба набора данных могут быть объединены, что значительно увеличивает количество используемых SNP.
Авторские взносы
CB-S, CM и YV разработали проект. CM, MC и JP выполнили молекулярную лабораторную работу. CB-S и J-BF проанализировали данные. CB-S и YV написали рукопись. Все авторы обсудили результаты и прокомментировали рукопись.
CM, MC и JP выполнили молекулярную лабораторную работу. CB-S и J-BF проанализировали данные. CB-S и YV написали рукопись. Все авторы обсудили результаты и прокомментировали рукопись.
Финансирование
Эта работа была поддержана грантом N ° ANR-12-PDOC-009-01 от Национального агентства исследований для CB-S, YV поддерживается грантом N ° ANR-13-BSV7-0017-01.
Заявление о конфликте интересов
Авторы заявляют, что исследование проводилось при отсутствии каких-либо коммерческих или финансовых отношений, которые могут быть истолкованы как потенциальный конфликт интересов.
Дополнительные материалы
Дополнительные материалы к этой статье можно найти в Интернете по адресу: https://www.frontiersin.org/article/10.3389/fpls.2016.007771
РИСУНОК S1 | Распределение частот минорных аллелей (MAF), оцененных в рамках конвейера TM для общих SNP.
ТАБЛИЦА S1 | Список SNP, общих для обоих наборов данных.
Сноски
- http://www.
 biotech.cornell.edu/brc/genomic-diversity
biotech.cornell.edu/brc/genomic-diversity - https://sites.google.com/site/africropproject/data
- http://www.R-project.org
Список литературы
Арнольд Б., Корбетт-Детиг Р. Б., Хартл Д. и Бомблис К. (2013). RADseq недооценивает разнообразие и вносит генеалогические ошибки из-за неслучайной выборки гаплотипов. Мол. Ecol. 22, 3179–3190. DOI: 10.1111 / mec.12276
PubMed Аннотация | CrossRef Полный текст | Google Scholar
Бэрд, Н. А., Эттер, П. Д., Этвуд, Т. С., Керри, М. К., Шивер, А. Л., Льюис, З. А. и др. (2008). Быстрое обнаружение SNP и генетическое картирование с использованием секвенированных маркеров RAD. PLoS ONE 3: e3376. DOI: 10.1371 / journal.pone.0003376
PubMed Abstract
SNP (однонуклеотидный полиморфизм) | DNAeXplained — Генетическая генеалогия
Вчера в журнале Nature была опубликована статья Маргаряна и др. «Популяционная геномика мира викингов», ставшая кульминацией 6-летней работы.
Всего несколько часов спустя Science Daily опубликовала статью: «Крупнейшее в мире секвенирование ДНК скелетов викингов показывает, что не все они были скандинавскими». Журнал Science опубликовал: «« Викинг »был описанием работы, а не вопросом наследственности, как показывают массовые исследования древней ДНК». Здесь писал National Geographic, здесь — CNN.
Викинги не все скандинавы или блондинки
Сказать что ??? Это совсем не то, что мы думали, что знаем. В науке самое замечательное — мы всегда узнаем что-то новое.
442 Скелеты викингов из-за пределов Скандинавии были секвенированы лабораторией Эске Виллерслева, в результате чего были получены последовательности полных геномов как для мужчин, так и для женщин из мест в Шотландии, Украине, Польше, России, Прибалтике, Исландии, Гренландии и других странах континентальной Европы. Затем их сравнили с известными образцами викингов из Скандинавии.
Не могила, из которой был взят образец, а кладбище викингов из Дании.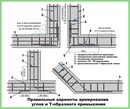
Одно захоронение в лодке викингов на кладбище викингов в Эстонии показывает, что 4 брата викингов умерли и были похоронены вместе, якобы погибшие в одном сражении, в один день.Судя по ДНК, братья, вероятно, приехали из Швеции.
Викинги совершают набеги из Скандинавии из Норвегии, Швеции и Дании. По крайней мере, некоторые налетчики викингов, похоже, тесно связаны друг с другом, а женщины в Исландии, похоже, с Британских островов, что позволяет предположить, что они, возможно, «стали» викингами — хотя мы не совсем понимаем социальную и общественную структуру.
Гена, обнаруженные у викингов, были внесены со всей Европы, включая южную Европу, и даже из Азии.Из-за смешения в результате набегов викингов, начавшихся в Линдисфарне в 793 году, население Великобритании сегодня несет до 6% ДНК викингов. Удивительно, но у шведов было всего 10%.
Некоторые захоронения викингов на Оркнейских островах и в Норвегии на самом деле были генетически пиктскими мужчинами.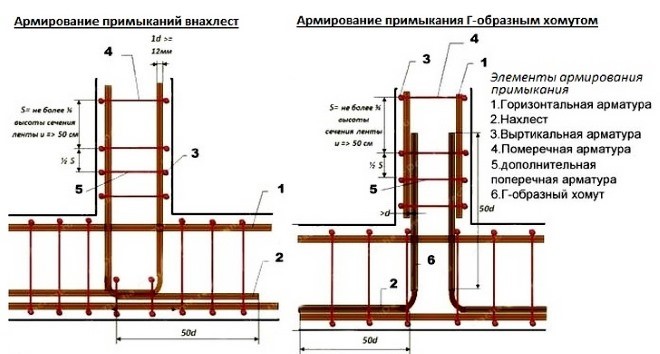 Возможно, обращаются? Одно из этих захоронений может быть самым ранним скелетом пиктов, обнаруженным на сегодняшний день.
Возможно, обращаются? Одно из этих захоронений может быть самым ранним скелетом пиктов, обнаруженным на сегодняшний день.
Y ДНК
Из 442 скелетов около 300 были мужчинами. Последовательность всего генома включает Y-хромосому вместе с митохондриальной ДНК, хотя для ее эффективного разделения требуется специальная обработка.
Горан Ранфельдт, член команды Million Mito и руководитель отдела исследований в FamilyTreeDNA, немедленно начал скачивать последовательности ДНК, а Майкл Сагер начал анализировать ДНК Y, надеясь добавить или разделить ветви Y ДНК дерева.
Учитывая недавнее разделение гаплогрупп P и A00, эти древние образцы обладают ОГРОМНЫМ обещанием.
Майкл и Горан согласились поделиться своей работой во время обработки этих образцов, что дает редкую возможность заглянуть в лабораторию в режиме реального времени.
Ты и дерево
Все в восторге от этой статьи, и я хочу, чтобы вы могли увидеть, соответствует ли ваша Y, митохондриальная ДНК или ДНК ваших родственников гаплогруппам ДНК, указанным в статье.
В самой статье для гаплогруппы ДНК Y используется старое буквенное обозначение, поэтому FamilyTreeDNA выполняет повторный запуск, выравнивание и сертификацию реальных SNP. Столбец FTDNA Haplogroup отражает имя гаплогруппы SNP Y.
Обратите внимание, что новые Y-ветви ДНК появляются на дереве в день ПОСЛЕ внесения изменений, и прямо сейчас изменения, связанные с этой статьей, вносятся ежечасно. Я буду обновлять информацию о гаплогруппах ежедневно по мере появления новых. Обратите особое внимание на места, которые показывают, где были найдены могилы, вместе с записями FamilyTreeDNA.
Горан также включил гаплогруппу мтДНК, как указано в статье. Гаплогруппы митохондриальной ДНК не пересчитывались, но вы можете увидеть их в Million Mito Project😊
Вот что вам нужно сделать:
- Перейдите к результатам анализа Y или митохондриальной ДНК и найдите свою гаплогруппу.
- Выполните поиск в браузере по этой статье, чтобы увидеть, отображается ли ваша гаплогруппа.
 На ПК это CTRL + F, чтобы отобразить поле поиска. Если ваша гаплогруппа не отображается, возможно, вы ниже по течению от гаплогруппы Viking, поэтому вам нужно будет использовать дерево блоков ДНК Y (для тестеров большого Y) или публичное гаплотерево здесь.
На ПК это CTRL + F, чтобы отобразить поле поиска. Если ваша гаплогруппа не отображается, возможно, вы ниже по течению от гаплогруппы Viking, поэтому вам нужно будет использовать дерево блоков ДНК Y (для тестеров большого Y) или публичное гаплотерево здесь. - Если вы прошли тест «Большой Y», нажмите «Дерево блоков» на странице результатов, а затем просмотрите верхнюю часть страницы результатов, чтобы узнать, является ли рассматриваемая гаплогруппа «вышестоящей» или родительской для вашей гаплогруппы.
нажмите для увеличения
Если вы его не видите, продолжайте сканирование влево, пока не увидите последний SNP.
нажмите для увеличения
- Если искомая гаплогруппа НЕ отображается в ваших прямых восходящих ветвях, вы можете ввести имя гаплогруппы в поле поиска.Например, я набрал I-BY3428. Вы также можете просто щелкнуть ссылку гаплогруппы имени FTDNA в таблице ниже, любезно предоставленную Гораном.
нажмите для увеличения
Я еще не вижу пересекающегося SNP между тестером и древним образцом, поэтому, если я нажму на I-Y2592, я могу просмотреть остальные вышестоящие ветви гаплогруппы I.
нажмите для увеличения
Глядя на SNP Y ДНК тестера и SNP Y ДНК древнего образца, я могу видеть, что пересекающийся SNP — это DF29, примерно 52 поколения SNP в прошлом.Эмпирическое правило состоит в том, что каждое поколение SNP составляет 80-100 лет.
- Если вы не тестировали свою ДНК Y и вы мужчина, вы можете заказать тест ДНК Y или перейти на Big Y-700 здесь. Если вы женщина, у которой нет ДНК Y, вы можете найти мужчину, чтобы проверить эту строку, или поискать в проектах фамилий, чтобы узнать, прошел ли кто-нибудь из вашей линии уже тестирование. Если это так, вы можете использовать публичное гаплотерево Y ДНК здесь. О том, как найти кандидатов на ДНК-тестирование, я писал здесь.
- Для митохондриальной ДНК вы можете использовать общедоступное гаплотрие митохондриальной ДНК здесь. Митохондриальная ДНК несколько проще, потому что она все еще написана в формате J1c1a, что означает, что J — корень, J1 — следующая ветвь, J1c — следующая младшая ветвь, а J1c1 — ветвь прямо над J1c1a.
 Если вы не заказывали тест митохондриальной ДНК, вы можете заказать или обновить его здесь.
Если вы не заказывали тест митохондриальной ДНК, вы можете заказать или обновить его здесь.
Как насчет вас — вы родственник викинга?
Ниже вы найдете информацию из результатов ДНК Y в документе, обработанную и проанализированную, с подтвержденными FamilyTreeDNA именами SNP, а также гаплогруппой митохондриальной ДНК каждого самца викингов.
Есть ли у вас родственники, и если да, то насколько тесно?
Я был удивлен, обнаружив сестринскую ветвь моей собственной митохондриальной J1c2f. J1c2 и несколько субкладов или ответвлений были найдены в захоронениях викингов.
Мне нужно проверить все мои родословные, как мужские, так и женские. Есть история, которая ждет своего раскрытия. Что ты открыл?
Образец информации о древних викингах
Обратите внимание, что эта информация будет обновляться в рабочие дни до тех пор, пока все образцы не будут обработаны и помещены в Y-дерево ДНК — так что это будет «живая» копия самой последней филогенетической информации.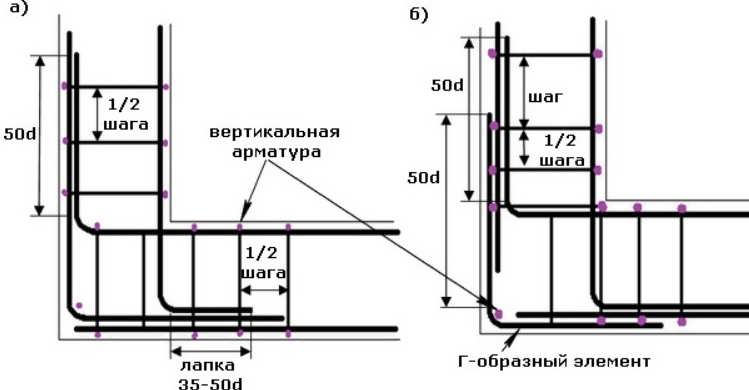
Ссылка на локации, чтобы увидеть локации раскопок и гаплогруппы локаций деревьев. Майкл Сагер комментирует каждый образец.
Наслаждайтесь!
Образец: VK14 / Russia_Ladoga_5680-12
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-BY3428
mtDNA: J1c1a
Образец: VK16 / Russia_Ladoga_5680-2
Местоположение: Ладога, Россия
Возраст: Викинг 11-12 вв. Н.э.
Y-ДНК: I-M253
мтДНК: X2b4
Образец: VK17 / Russia_Ladoga_5680-17
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: T-Y138678
FTDNA Комментарий: Делится 5 SNP с a человек из Чеченской Республики, формирующий новую ветвь вниз от T-Y22559 (T-Y138678)
мтДНК: U5a2a1b
Образец: VK18 / Россия_Ладога_5680-3
Расположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Н.э.
Y-ДНК: R-YP1370
мтДНК: h2b1
Образец: VK20 / Russia_Ladoga_5680-1
Местоположение: Ладога, Россия
Возраст: Викинг XI век н.э.
Y-ДНК: I-Y22478
FTDNA Комментарий: Разделяет ветвь I-Z24071, положительный только для Y22478.Новый путь = I-Y22486> I-Y22478> I-Z24071
мтДНК: H6c
Образец: VK22 / Russia_Ladoga_5680-13
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-A8462
мтДНК: T2b
Образец: VK23 / Russia_Ladoga_5680-9
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-M253
mtDNA: U4a1a
Образец: VK24 / Faroe_AS34 / Panum
Местоположение: Hvalba, Faroes
Возраст: Viking XI век
Y-ДНК: R-FGC12948
mtDNA: J1b1a1a
Образец: VK25 / Faroe_1
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н. э.
э.
Y-ДНК: R-FT381000
FTDNA Комментарий: Разделяет ветвь R-BY11762 , положительный для 5 вариантов, предков для ~ 14, новый путь = R-A8041> R-BY11764> BY11762
мтДНК: h4a1a
Образец: VK27 / Faroe_10
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-L513
мтДНК: U5a1g1
Образец: VK29 / Sweden_Skara 17
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: I-S7642
mtDNA: T2b3b
Образец: VK30 / Sweden_Skara 105
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 вв. Н. Э.
Y-ДНК: R-S2857
мтДНК: U5b1c2b
Образец: VK31 / Sweden_Skara 194
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н. э.
э.
Y-ДНК: R-L21
мтДНК: I4a
Образец: VK34 / Sweden_Skara 135
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: R-BY111759
mtDNA: HV-T16311C!
Образец: VK35 / Sweden_Skara 118
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-CTS4179
mtDNA: T2f1a1
Образец: VK39 / Sweden_Skara 181
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: G-Z1817
mtDNA: T2b4b
Образец: VK40 / Sweden_Skara 106
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-BY166438
FTDNA Комментарий: Делится 10 SNP с человек неизвестного происхождения (американец) после R-BY1701.Новая ветвь R-BY166438
мтДНК: T1a1
Образец: VK42 / Sweden_Skara 62
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н. э.
э.
Y-ДНК: J-FGC32685
mtDNA: T2b11
Образец: VK44 / Faroe_17
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S658
mtDNA: h4a1a
Образец: VK45 / Faroe_18
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-CTS8277
mtDNA: h4a1
Образец: VK46 / Faroe_19
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-BY202785
FTDNA Комментарий: Образует ветвь с VK245 вниз из R-BY202785 (Z287).Новая ветвь = R-FT383000
мтДНК: H5
Образец: VK48 / Gotland_Kopparsvik-212/65
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-FGC52679
mtDNA 9:
8 h20
Образец: VK50 / Gotland_Kopparsvik-53.64
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: I-Y22923
mtDNA: 9 hC2-T16
Образец: VK51 / Gotland_Kopparsvik-88/64
Расположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: N-L1026
mtDNA: U5b1e1
Образец: VK53 / Gotland_Kopparsvik-161/65
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: I-CTS10228
027 mtDNAb:8 HV9
Образец: VK57 / Gotland_Frojel-03601
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-L151
mtDNA: J1c6
Образец: VK60 / Gotland_Frojel-00702
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-YP1026
mtDNA: h23a1a1b
8
Образец: VK64 / Gotland_Frojel-03504
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-BY58559
mtDNA: I1a1
Образец: VK70 / Дания_Tollemosegard-EW
Местоположение: Толлемосегард, Силенд, Дания
Возраст: Ранний викинг Поздний германский железный век / ранний викинг
Y-ДНК: I-BY73576
mtDNA4:
Образец: VK71 / Дания_Tollemosegard-BU
Местоположение: Толлемосегард, Силенд, Дания
Возраст: Ранний викинг Поздний германский железный век / ранний викинг
Y-ДНК: I-S22349
mtDNA1:
Образец: VK75 / Гренландия, поздний 0929
Местоположение: V051, Западное поселение, Гренландия
Возраст: Поздний скандинавский 1300 г. н.э.
Y-ДНК: R-P310
мтДНК: H54
Образец: VK87 / Дания_Hesselbjerg Grav 41b, sk PC
Местоположение: Hesselbjerg, Ютландия, Дания
Возраст: Viking 850-900 CE
Y-ДНК: R-Z198
027 mtDNA2: K1
Образец: VK95 / Iceland_127
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: R-S658
mtDNA: H6a1a3a
Образец: VK98 / Iceland_083
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: I-BY3433
FTDNA Комментарий: Разделяет I-BY3430.Получено для 1 предка для 6. Новый путь = I-BY3433> I-BY3430
мтДНК: T2b3b
Образец: VK101 / Iceland_125
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-BY110718
мтДНК: U5b1g
Образец: VK102 / Iceland_128
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 веков н.э.
Y-ДНК: R-Y96503
FTDNA Комментарий: Делится 3 SNP с мужчиной из Швеция.Образует новую ветвь после R-FGC23826. Новая ветвь = R-Y96503
мтДНК: J1c3f
Образец: VK110 / Iceland_115S
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: I-FGC21682
мтДНК: h20-x
Образец: VK117 / Norway_Trondheim_SK328
Местоположение: Тронхейм, Нор-Мид, Норвегия
Возраст: Средневековье 12-13 века н.э.
Y-ДНК: R-S9257
мтДНК:
0 h2a3a 9 Образец: VK123 / Iceland_X104
Местоположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-Y130994
FTDNA Комментарий: Делится 17 SNP с мужчиной из ОАЭ.Создает новую ветвь после R2-V1180. Новая ветвь = R-Y130994
мтДНК: J1c9
Образец: VK127 / Iceland_HDR08
Местоположение: Хрингсдалур, Исландия
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY
мтДНК: h4g1b
Образец: VK129 / Iceland_ING08
Расположение: Ингиридарстадир, Исландия
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY154143
Комментарий FTDNA: Делится 3 SNP с мужчиной из Швеции.Образует новую ветвь после R1a-YP275. Новая ветвь = R-BY154143
мтДНК: U5b1b1a
Образец: VK133 / Дания_Galgedil KO
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 8-11 века н.э.
Y-ДНК: R-Z8
мтДНК: K1a4a1a3
Образец: VK134 / Дания_Galgedil ALZ
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY97519
мтДНК: h2cg
Образец: VK138 / Дания_Galgedil AQQ
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 век н.э.
Y-ДНК: R-S1491
мтДНК: T2b5
Образец: VK139 / Дания_Galgedil ANG
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32008
мтДНК: J1c3k
Образец: VK140 / Дания_Галгедил PT
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: G-M201
мтДНК: h37f
Образец: VK143 / UK_Oxford_ # 7
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y13833
FTDNA Комментарий: Разделяет R-Y13816.Получено для 6 предков для 3. Новый путь = R-Y13816> R-Y13833
мтДНК: U5b1b1-T16192C!
Образец: VK144 / UK_Oxford_ # 8
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-Y2592
mtDNA1:
Образец: VK145 / UK_Oxford_ # 9
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-YP1708
mtDNA:
Образец: VK146 / UK_Oxford_ # 10
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M6155
mtDNA3:
Образец: VK147 / UK_Oxford_ # 11
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y75899
mtDNA1q
Образец: VK148 / UK_Oxford_ # 12
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
mtDNA1a:
Образец: VK149 / UK_Oxford_ # 13
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
htDNA1:
Образец: VK150 / UK_Oxford_ # 14
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT4725
htDNA: mtDNA: -C16239T
Образец: VK151 / UK_Oxford_ # 15
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-S19291
mtDNA4: -T152C!
Образец: VK153 / Poland_Bodzia B1
Местоположение: Бодзя, Польша
Возраст: Викинг 10-11 века н.э.
Y-ДНК: R-M198
мтДНК: h2c3
Образец: VK156 / Poland_Bodzia B4
Расположение: Бодзия, Польша
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-Y9081
мтДНК: J1c2c2a
Образец: VK157 / Poland_Bodzia B5
Местоположение: Бодзия, Польша
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-S2077
мтДНК: h2c
Образец: VK159 / Russia_Pskov_7283-20
Местоположение: Псков, Россия
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-A7982
mtDNA: U2e2a1d
Образец: VK160 / Russia_Kurevanikka_7283-3
Местоположение: Куреваниха, Россия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: R-YP1137
мтДНК: C4a1a-T195C
Образец: VK163 / UK_Oxford_ # 1
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
mtDNA2 :a
Образец: VK165 / UK_Oxford_ # 3
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-S18218
mtDNA1:
Образец: VK166 / UK_Oxford_ # 4
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-BY67003
FTDNA Комментарий: Сплит R-BY45170 (DF27).Получено для 2, предков для 7. Новый путь = R-BY67003> R-BY45170
мтДНК: h4ag
Образец: VK167 / UK_Oxford_ # 5
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-BY34674
8 mtDNA1
Образец: VK168 / UK_Oxford_ # 6
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Z18
mtDNA1a mtDNA1a
Образец: VK170 / Isle-of-Man_Balladoole
Местоположение: Balladoole, IsleOfMan
Возраст: Викинг 9-10 веков н.э.
Y-ДНК: R-S3201
mtDNA:
05 HV9b 9b 9b Образец: VK172 / UK_Oxford_ # 16
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT7019
mtDNA1:
Образец: VK173 / UK_Oxford_ # 17
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT13004
FTDNA Комментарий: Расщепляет I2-FT12648, полученный для 5, наследственный для 7.Новый путь FT13004> FT12648
мтДНК: U5a1b-T16362C
Образец: VK174 / UK_Oxford_ # 18
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FGC17429
mtDNA -C16239T
Образец: VK175 / UK_Oxford_ # 19
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y47841
FTDNA Комментарий: Делит 6 SNP с человеком из Швеции, ниже R-BY38950 (R-Y47841)
мтДНК: h2a1
Образец: VK176 / UK_Oxford_ # 20
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT3562
mtDNA: mtDNA:
Образец: VK177 / UK_Oxford_ # 21
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT31867
FTDNA Комментарий: Делится 3 SNP с мужчиной из Греции.Образует новую ветвь после R-BY220332 (U152). Новая ветвь = R-FT31867
мтДНК: H82
Образец: VK178 / UK_Oxford_ # 22
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-BY176639
FTDNA Комментарий: Соединяется с PGA3 (Проект персонального генома, Австрия) и клиентом FTDNA из Дании. Клиенты PGA и FTDNA сформировали филиал ранее на этой неделе, VK178 присоединится к ним по адресу R-BY176639 (под L48)
мтДНК: K2a5
Образец: VK179 / Гренландия F2
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Раннее скандинавское население 10–12 веков н. Э.
Y-ДНК: I-F3312
мтДНК: K1a3a
Образец: VK183 / Гренландия F6
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н.э.
Y-ДНК: I-F3312
мтДНК: T2b21
Образец: VK184 / Гренландия F7
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н. Э.
Y-ДНК: R-YP4342
мтДНК: h5a1a4b
Образец: VK186 / Гренландия KNK- [6]
Местоположение: Ø64, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 века н.э.
Y-ДНК: I-Y79817
FTDNA Комментарий : Делится 3 SNP с мужчиной из Норвегии ниже I-Y24625.Новая ветвь = I-Y79817
мтДНК: h2ao
Образец: VK190 / Гренландия, поздний-0996
Местоположение: Ø149, Восточное поселение, Гренландия
Возраст: Поздний скандинавский 1360 г. н.э.
Y-ДНК: I-FGC15543
FTDNA Комментарий: Сплит I- FGC15561. Получено 11 предков для 6. Новый путь = I-FGC15543> I-FGC15561
мтДНК: K1a-T195C!
Образец: VK201 / Orkney_Buckquoy, sk M12
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Викинг 5-6 века н.э.
Y-ДНК: I-B293
mtDNA1
Образец: VK202 / Orkney_Buckquoy, sk 7B
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Viking 10 век н.э.
Y-ДНК: R-A151
mtDNA1
Образец: VK203 / Orkney_BY78, Ar.1, sk 3
Расположение: Brough_Road_Birsay, Оркни, Шотландия, Великобритания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY10450
FTDNA Комментарий: FT83323-
0 mtDNA1 9a a h5a
Образец: VK204 / Orkney_Newark for Brothwell
Местоположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY115469
mtDNA 9:
8 h2
Образец: VK205 / Orkney_Newark 68/12
Расположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-YP4345
mtDNA:
8 h4
Образец: VK210 / Poland_Kraków-Zakrzówek gr.24
Местоположение: Краков, Польша
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: I-Z16971
мтДНК: H5e1a1
Образец: VK211 / Польша_Cedynia gr. 435
Местоположение: Cedynia, Poland
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: R-M269
мтДНК: W6
Образец: VK212 / Польша_Cedynia gr. 558
Расположение: Сединия, Польша
Возраст: Викинг 11-12 века н.э.
Y-ДНК: R-CTS11962
мтДНК: h2-T152C!
Образец: VK215 / Дания_Гердруп-Б; sk 1
Местоположение: Гердруп, Силенд, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-M269
мтДНК: J1c2k
Образец: VK217 / Sweden_Ljungbacka
Местоположение: Ljungbacka, Мальме, Швеция
Возраст: Викинг 9-12 вв. Н.э.
Y-ДНК: R-L151
мтДНК: J1b1b1
Образец: VK218 / Russia_Ladoga_5680-4
Расположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: R-BY2848
мтДНК: H5
Образец: VK219 / Russia_Ladoga_5680-10
Местоположение: Ладога, Россия
Возраст: Викинг 10-11 века н.э.
Y-ДНК: I-Y22024
mtDNA: T2b6a
Образец: VK220 / Russia_Ladoga_5680-11
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-FT253975
FTDNA Комментарий: CTS2208 +, BY47171-, CTS7676-, Y20288-, BY69785-, FT253975 +
мтДНК: J2b1a
Образец: VK221 / Russia_Ladoga_5757-14
Местоположение: Ладога, Россия
Возраст: Викинг 9-10 вв. Н.э.
Y-ДНК: I-Y5473
мтДНК: K1d
Образец: VK223 / Россия_Гнездово 75-140
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: I-BY67763
мтДНК: h23a1a1c
Образец: VK224 / Россия_Гнездово 78-249
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: N-CTS2929
мтДНК: H7a1
Образец: VK225 / Iceland_A108
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-BY
мтДНК: h4v-T16093C
Образец: ВК232 / Готланд_Коппарсвик-240.65
Расположение: Коппарсвик, Готланд, Швеция
Возраст: Викинг 900-1050 CE
Y-ДНК: R-Y16505
FTDNA Комментарий: Спекулятивное размещение — U106 +, но U106 (C> T) в древние образцы могут ввести в заблуждение. LAV010, NA34, I7779, ble007, R55 и EDM124 — все это древние образцы, не относящиеся к R и являющиеся U106 +. Более консервативное размещение — R-P310
мтДНК: N1a1a1
Образец: VK234 / Faroe_2
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-FT381000
FTDNA Комментарий: То же разделение, что и VK25.У них общий маркер FT381000 (26352237 T> G)
мтДНК: h4a1a
Образец: VK237 / Faroe_15
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S6355
mtDNA: J2a2c
Образец: VK238 / Faroe_4
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-YP396
mtDNA: h4a1a
Образец: VK239 / Faroe_5
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-M269
mtDNA: H5
Образец: VK242 / Faroe_3
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S764
mtDNA: h4a1a
Образец: VK244 / Faroe_12
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-CTS4179
mtDNA: h3a2a2
Образец: VK245 / Faroe_16
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-BY202785
FTDNA Комментарий: Образует ветвь с VK46 вниз из R-BY202785 (Z287).Новая ветвь = R-FT383000
мтДНК: h4a1
Образец: VK248 / Faroe_22
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: I-M253
мтДНК: h59a
Образец: VK251 / Gotland_Kopparsvik-30.64
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-M459
mtDNA: 9 U5b1e
Образец: VK256 / UK_Dorset-3722
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-YP5718
m
Образец: VK257 / UK_Dorset-3723
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-Y19934
ma
Образец: VK258 / UK_Dorset-3733
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-YP1395
FTDNA Комментарий: FTDNA 5 SNP с мужчиной из Норвегии.Образует новую ветвь R-YP1395. Новая ветвь = R-Ph520
мтДНК: K1a4a1
Образец: VK259 / UK_Dorset-3734
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-FT20255
Оба VK449 и VK259 делят 3 SNP с мужчиной из Швеции. Образует новую ветвь вниз R-FT20255 (Z18). Новая ветвь = R-FT22694
мтДНК: I2
Образец: VK260 / UK_Dorset-3735
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК:
0t Q-BY77336
m Образец: VK261 / UK_Dorset-3736
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-BY64643
Y-ДНК: R-BY64643
m
Образец: VK262 / UK_Dorset-3739
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-FT347811
FTDNA Комментарий: FT347811
2 SNP с американцем неизвестного происхождения.Образует новую ветвь вниз от Y6908 (Z140). В то же время была обнаружена новая ветвь, которая объединяет эту новую древнюю / американскую ветвь с установленной веткой I-FT274828. Новый древний путь = I-Y6908> I-FT273257> I-FT347811
мтДНК: J1c4
Образец: VK263 / UK_Dorset-3742
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: K-Z16372
m
Образец: VK264 / UK_Dorset-3744
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Образец: VK267 / Sweden_Karda 21
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-L23
мтДНК: T2b4b
Образец: VK268 / Sweden_Karda 22
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
мтДНК: K1c1
Образец: VK269 / Sweden_Karda 24
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
mtDNA: h2e1a
Образец: VK273 / Россия_Гнездово 77-255
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: R-BY61747
мтДНК: U5a2a1b1
Образец: VK274 / Дания_Kaargarden 391
Местоположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-Ph4519
mtDNA: T2b-T152C!
Образец: VK275 / Дания_Kaargarden 217
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY74743
мтДНК: H
Образец: VK279 / Дания_Galgedil AX
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y10639
мтДНК: I4a
Образец: VK280 / Дания_Galgedil UO
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y3713
мтДНК: h21a
Образец: VK281 / Дания_Barse Grav A
Местоположение: Борсе, Силенд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-FGC22153
Комментарий FTDNA: Разделяет I-Y5612 ( P109).Получено для 8, предков для 2. Новый путь = I-Y5612> I-Y5619
мтДНК: T2
Образец: VK282 / Дания_Stengade I, LMR c195
Местоположение: Stengade_I, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-CTS1211
mtDNA: 8 h5a
Образец: VK286 / Дания_Bogovej Grav BJ
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-S10708
mtDNA: J1c-C16261
Образец: VK287 / Дания_Kaargarden Grav BS
Местоположение: Kaagården, Langeland, Дания
Возраст: Viking X век н.э.
Y-ДНК: R-S22676
mtDNA: T2b
Образец: VK289 / Дания_Bodkergarden Grav H, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-U106
mtDNA:
8 J2b1a
Образец: VK290 / Дания_Kumle Hoje Grav O
Местоположение: Kumle_høje, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-FT264183
FTDNA Комментарий: Акционирует не менее SNP с мужчиной из Швеции, образуя новую ветвь ниже по течению R-FT263905 (U106).Новая ветка = R-FT264183. HG02545 остается в R-FT263905
мтДНК: I1a1
Образец: VK291 / Дания_Bodkergarden Grav D, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг, 9 век н.э.
Y-ДНК: I-Y20861
mtDNA: U5a
Образец: VK292 / Дания_Bogovej Grav A.D.
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-M417
мтДНК:
028 J1c2c1 9 Образец: VK295 / Дания_Hessum sk 1
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y4738
мтДНК: T1a1
Образец: VK296 / Дания_Hundstrup Mose sk 1
Расположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 660-780 CE
Y-ДНК: I-S7660
mtDNA: HV6
Образец: VK297 / Дания_Hundstrup Mose sk 2
Местоположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 670-830 CE
Y-ДНК: I-Y4051
mtDNA:
0 J1c2h 9 Образец: VK301 / Дания_Ladby Grav 4
Местоположение: Ladby, Funen, Дания
Возраст: Viking 640-890 CE
Y-ДНК: I-FT105192
mtDNA: R0a2b
Образец: VK306 / Sweden_Skara 33
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: I-FT115400
FTDNA Комментарий: Имеет 3 мутации с мужчина из Швеции.Образует новую ветвь I-S19291. Новая ветка = I-FT115400. VK151 не имеет покрытия для 2 из этих мутаций
мтДНК: h25a1
Образец: VK308 / Sweden_Skara 101
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: R-BY33037
мтДНК: h2c
Образец: VK309 / Sweden_Skara 53
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-YP6189
мтДНК: K1b1c
Образец: VK313 / Дания_Rantzausminde Grav 2
Местоположение: Ранцаусминде, Фюн, Дания
Возраст: Викинг 850-900 CE
Y-ДНК: R-JFS0009
мтДНК: h2b
Образец: VK315 / Дания_Bakkendrup Grav 16
Местоположение: Баккендруп, Силенд, Дания
Возраст: Viking 850-900 CE
Y-ДНК: I-Y98280
Комментарий FTDNA: Делится 1 SNP с мужчина из Нидерландов.Образует новую ветвь после I-Y37415 (P109). Новая ветвь = I-Y98280
мтДНК: T1a1b
Образец: VK316 / Дания_Hessum sk II
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y130659
Комментарий FTDNA: Сплит I- Y130594 (Z59). Получено для 1 предка для 6. Новый путь = I-Y130659> I-Y130594> I-Y130747. Древний образец STR_486 также принадлежит к этой группе, по I-Y130747
мтДНК: K1a4
Образец: VK317 / Дания_Kaargarden Grav BF99
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: J-BY62479
FTDNA Комментарий: 6-Splits J2 M67).Получено для 9, предков для 3. Новый путь = J-BY62479> J-BY72550
мтДНК: h3a2a1
Образец: VK320 / Дания_Bogovej Grav S
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y103013
Комментарий FTDNA: Делится 3 SNP с мужчина из Швеции. Образует новую ветвь вниз I-FT3562 (P109). Новая ветвь = I-Y103013
мтДНК: U5a1a1
Образец: VK323 / Дания_Рибе 2
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-S10185
мтДНК: K2a6
Образец: VK324 / Дания_Ribe 3
Расположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY16590
FTDNA Комментарий: Сплит R-BY16590 (L47).Получено для 7, предков для 3. Новый путь = R-S9742> R-BY16950
мтДНК: N1a1a1a2
Образец: VK326 / Дания_Рибе 5
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-Y52895
mtDNA: U5b1-T16189C! — T16192C!
Образец: VK327 / Дания_Рибе 6
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-BY463
мтДНК: H6a1a5
Образец: VK329 / Дания_Ribe 8
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинги 9-11 веков н.э.
Y-ДНК: R-S18894
мтДНК: h4-T152C!
Образец: VK332 / Oland_1088
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-S8522
FTDNA Комментарий: Возможно, ниже I-BY195155.Имеет одну мутацию C> T с образцом BY195155 *
мтДНК: T2b24
Образец: VK333 / Oland_1028
Местоположение: Эланд, Швеция
Возраст: Viking 885 ± 69 CE
Y-ДНК: R-Z29034
mtDNA: h3a2a1
Образец: VK335 / Oland_1068
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н. Э.
Y-ДНК: R-BY39347
FTDNA Комментарий: Делится 8 SNP с мужчиной Франция.Образует новую ветвь вниз по R-BY39347 (U152). Новая ветвь = R-FT304388
мтДНК: K1b2a3
Образец: VK336 / Oland_1075
Местоположение: Эланд, Швеция
Возраст: Viking 853 ± 67 CE
Y-ДНК: R-BY106906
мтДНК: K2a3a
Образец: VK337 / Oland_1064
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-BY31739
FTDNA Комментарий: Возможный Z140
mtDNA1b: 9003a U5a
Образец: VK338 / Дания_Bogovej Grav BV
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-A6707
мтДНК: W3a1
Образец: VK342 / Oland_1016
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-BY78615
FTDNA Комментарий: Имеет 2 SNP с мужчиной из Финляндия.Образует новую ветвь вниз I2-Y23710 (L801). Новая ветвь = I-BY78615
мтДНК: h3a1
Образец: VK343 / Oland_1021
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y7232
мтДНК: h4h
Образец: VK344 / Oland_1030
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32357
mtDNA: J1c2t
Образец: VK345 / Oland_1045
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-FT148754
FTDNA Комментарий: Сплит R-FT148754 (DF63) .Получено для 8, предков для 6. Новый путь = R-FT148796> R-FT148754
мтДНК: h5a1
Образец: VK346 / Oland_1057
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 века н.э.
Y-ДНК: J-Z8424
мтДНК: h3a2b
Образец: VK348 / Oland_1067
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Z171
мтДНК: T2b28
Образец: VK349 / Oland_1073
Местоположение: Оланд, Швеция
Возраст: Viking 829 ± 57 CE
Y-ДНК: R-BY166065
FTDNA Комментарий: Делится 2 SNP с мужчиной из Англии .Образует ответвление вниз от R-BY166065 (L1066). Новая ветвь = R-BY167052
мтДНК: h2e2a
Образец: VK352 / Oland_1012
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-FGC35755
FTDNA Комментарий: Возможно, образует ветвь вниз I -Y15295. 2 возможные мутации G> A с образцом I-Y15295 *
мтДНК: H64
Образец: VK354 / Oland_1026
Местоположение: Эланд, Швеция
Возраст: Viking 986 ± 38 CE
Y-ДНК: R-S6752
мтДНК: h3a1
Образец: VK355 / Oland_1046
Местоположение: Эланд, Швеция
Возраст: Викинг 847 ± 65 CE
Y-ДНК: L-L595
FTDNA Комментарий: Присоединяется к 2 другим древним на этой редкой ветви .ASH087 и I2923
мтДНК: U5b1b1a
Образец: VK357 / Oland_1097
Местоположение: Эланд, Швеция
Возраст: Viking 1053 ± 60 CE
Y-ДНК: I-FT49567
FTDNA Комментарий: Делится 4 SNP с мужчиной из Англии . Образует новую ветвь I-A5952 (Z140). Новая ветвь = I-FT49567
мтДНК: J2b1a
Образец: VK362 / Дания_Bogovej LMR 12077
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: E-CTS5856
FTDNA Комментарий:
3 Возможно 90 E-Z мтДНК: V7b
Образец: VK363 / Дания_Bogovej BT
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY198083
Комментарий FTDNA: Делится 2 SNP с мужчиной из Швейцарии.Образует новую ветвь I-A1472 (Z140). Новая ветвь = I-BY198083
мтДНК: U4b1a1a1
Образец: VK365 / Дания_Bogovej BS
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY34800
мтДНК: U8a2
Образец: VK367 / Дания_Bogovej D
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY67827
Комментарий FTDNA: VK506 и VK19967 9 Внутренние отделы — SCP Foundation
Успешная работа фонда зависит, прежде всего, от разделения ответственности между различными отделами.Некоторые подразделения Фонда перечислены в этом документе. Ради эффективности все отделы тесно координируются.
Административный отдел
Административный отдел возглавляют непосредственно команда O5 и администратор. Он направляет действия Фонда на всех уровнях. В этот отдел входят все узлы связи и командные центры; у каждого такого узла и центра есть резерв на случай отказа.В юрисдикцию AD также входят финансовые операции Фонда в целом, такие как распоряжение средствами, бухгалтерский учет, планирование бюджета и т. Д. Из-за важности этого отдела проверки лояльности здесь проводятся чаще, чем где-либо еще. К этому же отделу относится делопроизводство. Департамент иностранных дел (DEA)
DEA отвечает за широкий спектр операций: распространение дезинформации, устранение следов деятельности SCP (как объектов, так и деятельности Фонда), набор персонала класса D и набор новых сотрудников из военных и гражданских учреждений.DEA отвечает за сокрытие деятельности агентов и объектов Фонда в населенных пунктах. DEA — это, по сути, передовая линия Фонда, поскольку она фильтрует всех потенциальных сотрудников и отвечает за поддержание высоких стандартов среди нанятого персонала — это также можно назвать HR. Отдел инженерно-технического обслуживания
E&TS отвечает за техническое обслуживание и повседневное обслуживание всех объектов и оборудования, активно используемых Фондом.Этот отдел отвечает за проектирование и строительство новых объектов и путей сообщения, а также за ремонт и техническое обслуживание. Отдел E&TS разделен на несколько подотделов в зависимости от уровней допуска и ассигнований; сотрудники с более высоким уровнем допуска проектируют и строят камеры содержания класса Кетер и работают со стратегически важными объектами. Комитет по этике
Комитет по этике — небольшой независимый отдел, отвечающий за рассмотрение процедур сдерживания и проверку проведенных экспериментов на предмет чрезмерного расточительства или ненадлежащего использования сотрудников класса D и других ресурсов.Некоторые считают существование комитета плохой шуткой, но он играет значительную роль в деятельности Фонда и имеет большое влияние. Разведывательное управление
Разведывательное управление занимается поиском, отслеживанием и захватом несохраняемых объектов SCP, а также сбором разведданных о враждебных группах интересов. Ячейки под прикрытием IA существуют во всех крупных населенных пунктах и замаскированы под гражданские объекты и учреждения.Агенты разведки обычно набираются из различных силовых структур и спецслужб мира. Отдел внутренней безопасности
ISD — это скрытый «фонд в Фонде», силы тайной полиции, ответственные за фильтрацию предателей, а также за оперативные риски и риски информационной безопасности в рядах Фонда. Отдел строго иерархичен, придерживаясь строго определенного многоэтапного протокола расследования.Потенциальные агенты ISD должны иметь безупречный стаж работы и стаж работы в Фонде более года. Обычно каждый агент ISD сохраняет свою «официальную» позицию, чтобы предоставить своей команде отчет из первых рук обо всем, что происходит в данном отделе. Само существование ISD обычно преподносится как миф. «Официальная» ответственность ИСД — это расследование и допрос захваченных агентов ГОИ, хотя эта часть ИСД официально принадлежит Управлению безопасности в целях секретности. Отдел логистики
Департамент логистики отвечает за передачу природных, финансовых и человеческих ресурсов между объектами Фонда. Транспортировка осуществляется автомобильным, водным, железнодорожным, воздушным, космическим и другим транспортом. Имеется сложная система распределения приоритетов и классов важности перевозок; Объекты SCP и ресурсы, необходимые для их сдерживания, обычно получают высший приоритет.Устранение препятствий, возникающих при транспортировке, также является первоочередной задачей. Производственный отдел
Производственный отдел отвечает за выполнение любых материальных запросов, которые могут возникнуть во время поимки, содержания или любого другого процесса Фонда. Производственный отдел имеет несколько участков и зон, полностью посвященных созданию широкого профиля изделий на заказ или серийного производства. Проектирование и производство часто осуществляется на месте по соображениям безопасности.По тем же причинам ни производственный персонал, ни проектная документация никогда не покидают пределы объекта. Обычно они оснащены современным оборудованием и тщательно охраняются. Лечебное отделение
Медицинский отдел Фонда отвечает за поддержание хорошего здоровья сотрудников. Из-за характера работы Фонда медики часто сталкиваются с трудно поддающимися лечению и весьма необычными случаями ран, травм, инфекций и расстройств.В связи с этим ОД придерживается исключительно высоких кадровых стандартов. Некоторые медики Фонда специализируются на вторичных областях лечения, таких как меметические воздействия, психологические травмы и т. Д. Мобильные оперативные группы
Работа Фонда требует большого количества узкоспециализированных специалистов. Обычно это связано с аномалиями, которые требуют больших усилий и нестандартного подхода к локализации.Для решения этой проблемы создаются мобильные оперативные группы. MTF — это группы разного размера, которые специализируются на определенных ролях или реагируют на конкретные ситуации. У каждой группы есть название, состоящее из греческой буквы и числа; уточняющие префиксы и / или позывные могут использоваться в некоторых случаях. Дополнительную информацию можно найти в Обзоре мобильных оперативных групп. Научный отдел
Эффективная научная работа — краеугольный камень существования и деятельности Фонда.Научный отдел отвечает за изучение всех содержащихся и новых SCP-объектов, разработку мер противодействия событиям нарушения условий содержания и разработку различных устройств и лекарственных препаратов, которые будут производиться Производственным отделом. Сотрудники научного отдела набираются из числа лучших и перспективных ученых в различных областях. Отдел безопасности
Этот отдел отвечает за обеспечение безопасности и защиты всех помещений и объектов Фонда, независимо от их приоритета.Сотрудники SD обучены противодействию событиям нарушения условий содержания, вторжениям, саботажу, стихийным бедствиям и другим чрезвычайным ситуациям. Новички в этой услуге обычно проходят общее обучение перед тем, как попасть на объект или площадку. Специальное обучение работе с данным объектом проводится на выезде после задания. Департамент внутреннего трибунала
Отдел внутреннего трибунала Фонда SCP является институциональным судебным органом организации.Этот отдел, по просьбе ISD, рассматривает дела сотрудников Фонда и других лиц, которые подпадают под определение запрещенных Правовым кодексом Департамента внутреннего трибунала, включая такие записи, как: «I — Преступления против Фонда», «II — Преступления против нормальности и человечности »,« III — Существенные дисциплинарные нарушения »,« IV — Подготовка к проведению мероприятия класса K »,« V — Преступление государства и корпорации »,« VI — Прочие нарушения ». Трибунал может назначить различные приговоры по каждой категории нарушений (за исключением пятого, судебное разбирательство по которому ведется ГОК).Любое решение Трибунала может быть отменено Командованием O5. Вы можете ознакомиться с Положением о внутреннем трибунале Фонда SCP и его Юридическим кодексом здесь. Центральные базы данных мутаций и SNP
Репозиторий dbSNP-полиморфизмов NCBI, NIH, Bethesda Сервер молекулярной биологии ExPASy Швейцарский институт биоинформатики (SIB) Европейский институт биоинформатики EBI EMBL / EBI GeneSNPs (общедоступный Интернет-ресурс) Команда Центра генома Университета Юты ClinVar, NCBI Национальный центр биотехнологической информации Генатлас Жан Фрезаль, INSERM, Генетон, Франция, База данных генных карт М.Ребхан, Дж. Прилуски, В. Чалифа-Каспи и Дорон Ланцет Институт Вейцмана, Реховот, Израиль GVS: сервер изменения генома Д-р Дебора Никерсон, д-р Марк Ридер и команда США HGVbase: база данных вариаций генома человека Каролинский институт, Стокгольм, Швеция Европейский институт биоинформатики (EBI), Хинкстон, Великобритания и Европа, лаборатория молекулярной биологии (EMBL), Гейдельберг, Германия HOWDY: Организованная база данных полного генома человека Японская научно-техническая корпорация База данных мутаций генов человека, HGMD Д.Н. Купер, Э. В. Болл, П. Д. Стенсон М. Кравчак, Univ. Медицинского колледжа Уэльса. Кардифф, Великобритания jSNP: база База данных японских SNP Центр генома человека, Институт медицинских наук, Токийский университет База данных SNP Leelab Иризарри, К. Кустанович, В. Ли, К.Браун, Н. Нельсон, С. Вонг, В., Ли, К.Дж. UCLA Центр биоинформатики База данных человеческих SNP Институт Уайтхеда / Центр исследований генома Массачусетского технологического института Интернет-Менделирующее наследование в человеке, OMIM В. А. МакКусик и его коллеги, Университет Джона Хопкинса, NCBI NIEHS SNP ДокторДебора Никерсон, доктор Марк Ридер и команда США PharmGKB: База знаний по фармакогенетике и фармакогеномике Стэнфордский университет США SNP в Сиэтле Д-р Дебора Никерсон, д-р Марк Ридер и их команда Вашингтонского университета, Принстонского университета и Национального института сердца, легких и крови, U.
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-L151
mtDNA: J1c6
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-YP1026
mtDNA: h23a1a1b
Местоположение: Frojel, Gotland, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-BY58559
mtDNA: I1a1
Местоположение: Толлемосегард, Силенд, Дания
Возраст: Ранний викинг Поздний германский железный век / ранний викинг
Y-ДНК: I-BY73576
mtDNA4:
Местоположение: Толлемосегард, Силенд, Дания
Возраст: Ранний викинг Поздний германский железный век / ранний викинг
Y-ДНК: I-S22349
mtDNA1:
Местоположение: V051, Западное поселение, Гренландия
Возраст: Поздний скандинавский 1300 г. н.э.
Y-ДНК: R-P310
мтДНК: H54
Местоположение: Hesselbjerg, Ютландия, Дания
Возраст: Viking 850-900 CE
Y-ДНК: R-Z198
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: R-S658
mtDNA: H6a1a3a
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: I-BY3433
FTDNA Комментарий: Разделяет I-BY3430.Получено для 1 предка для 6. Новый путь = I-BY3433> I-BY3430
мтДНК: T2b3b
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-BY110718
мтДНК: U5b1g
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 веков н.э.
Y-ДНК: R-Y96503
FTDNA Комментарий: Делится 3 SNP с мужчиной из Швеция.Образует новую ветвь после R-FGC23826. Новая ветвь = R-Y96503
мтДНК: J1c3f
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: I-FGC21682
мтДНК: h20-x
Местоположение: Тронхейм, Нор-Мид, Норвегия
Возраст: Средневековье 12-13 века н.э.
Y-ДНК: R-S9257
мтДНК:
Образец: VK123 / Iceland_X104
Местоположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-Y130994
FTDNA Комментарий: Делится 17 SNP с мужчиной из ОАЭ.Создает новую ветвь после R2-V1180. Новая ветвь = R-Y130994
мтДНК: J1c9
Образец: VK127 / Iceland_HDR08
Местоположение: Хрингсдалур, Исландия
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY
мтДНК: h4g1b
Образец: VK129 / Iceland_ING08
Расположение: Ингиридарстадир, Исландия
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY154143
Комментарий FTDNA: Делится 3 SNP с мужчиной из Швеции.Образует новую ветвь после R1a-YP275. Новая ветвь = R-BY154143
мтДНК: U5b1b1a
Образец: VK133 / Дания_Galgedil KO
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 8-11 века н.э.
Y-ДНК: R-Z8
мтДНК: K1a4a1a3
Образец: VK134 / Дания_Galgedil ALZ
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY97519
мтДНК: h2cg
Образец: VK138 / Дания_Galgedil AQQ
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 век н.э.
Y-ДНК: R-S1491
мтДНК: T2b5
Образец: VK139 / Дания_Galgedil ANG
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32008
мтДНК: J1c3k
Образец: VK140 / Дания_Галгедил PT
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: G-M201
мтДНК: h37f
Образец: VK143 / UK_Oxford_ # 7
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y13833
FTDNA Комментарий: Разделяет R-Y13816.Получено для 6 предков для 3. Новый путь = R-Y13816> R-Y13833
мтДНК: U5b1b1-T16192C!
Образец: VK144 / UK_Oxford_ # 8
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-Y2592
mtDNA1:
Образец: VK145 / UK_Oxford_ # 9
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-YP1708
mtDNA:
Образец: VK146 / UK_Oxford_ # 10
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M6155
mtDNA3:
Образец: VK147 / UK_Oxford_ # 11
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y75899
mtDNA1q
Образец: VK148 / UK_Oxford_ # 12
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
mtDNA1a:
Образец: VK149 / UK_Oxford_ # 13
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
htDNA1:
Образец: VK150 / UK_Oxford_ # 14
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT4725
htDNA: mtDNA: -C16239T
Образец: VK151 / UK_Oxford_ # 15
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-S19291
mtDNA4: -T152C!
Образец: VK153 / Poland_Bodzia B1
Местоположение: Бодзя, Польша
Возраст: Викинг 10-11 века н.э.
Y-ДНК: R-M198
мтДНК: h2c3
Образец: VK156 / Poland_Bodzia B4
Расположение: Бодзия, Польша
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-Y9081
мтДНК: J1c2c2a
Образец: VK157 / Poland_Bodzia B5
Местоположение: Бодзия, Польша
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-S2077
мтДНК: h2c
Образец: VK159 / Russia_Pskov_7283-20
Местоположение: Псков, Россия
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-A7982
mtDNA: U2e2a1d
Образец: VK160 / Russia_Kurevanikka_7283-3
Местоположение: Куреваниха, Россия
Возраст: Викинг 10-13 века н.э.
Y-ДНК: R-YP1137
мтДНК: C4a1a-T195C
Образец: VK163 / UK_Oxford_ # 1
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-M253
mtDNA2 :a
Образец: VK165 / UK_Oxford_ # 3
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-S18218
mtDNA1:
Образец: VK166 / UK_Oxford_ # 4
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-BY67003
FTDNA Комментарий: Сплит R-BY45170 (DF27).Получено для 2, предков для 7. Новый путь = R-BY67003> R-BY45170
мтДНК: h4ag
Образец: VK167 / UK_Oxford_ # 5
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-BY34674
8 mtDNA1
Образец: VK168 / UK_Oxford_ # 6
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Z18
mtDNA1a mtDNA1a
Образец: VK170 / Isle-of-Man_Balladoole
Местоположение: Balladoole, IsleOfMan
Возраст: Викинг 9-10 веков н.э.
Y-ДНК: R-S3201
mtDNA:
05 HV9b 9b 9b Образец: VK172 / UK_Oxford_ # 16
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT7019
mtDNA1:
Образец: VK173 / UK_Oxford_ # 17
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT13004
FTDNA Комментарий: Расщепляет I2-FT12648, полученный для 5, наследственный для 7.Новый путь FT13004> FT12648
мтДНК: U5a1b-T16362C
Образец: VK174 / UK_Oxford_ # 18
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FGC17429
mtDNA -C16239T
Образец: VK175 / UK_Oxford_ # 19
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y47841
FTDNA Комментарий: Делит 6 SNP с человеком из Швеции, ниже R-BY38950 (R-Y47841)
мтДНК: h2a1
Образец: VK176 / UK_Oxford_ # 20
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT3562
mtDNA: mtDNA:
Образец: VK177 / UK_Oxford_ # 21
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT31867
FTDNA Комментарий: Делится 3 SNP с мужчиной из Греции.Образует новую ветвь после R-BY220332 (U152). Новая ветвь = R-FT31867
мтДНК: H82
Образец: VK178 / UK_Oxford_ # 22
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-BY176639
FTDNA Комментарий: Соединяется с PGA3 (Проект персонального генома, Австрия) и клиентом FTDNA из Дании. Клиенты PGA и FTDNA сформировали филиал ранее на этой неделе, VK178 присоединится к ним по адресу R-BY176639 (под L48)
мтДНК: K2a5
Образец: VK179 / Гренландия F2
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Раннее скандинавское население 10–12 веков н. Э.
Y-ДНК: I-F3312
мтДНК: K1a3a
Образец: VK183 / Гренландия F6
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н.э.
Y-ДНК: I-F3312
мтДНК: T2b21
Образец: VK184 / Гренландия F7
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н. Э.
Y-ДНК: R-YP4342
мтДНК: h5a1a4b
Образец: VK186 / Гренландия KNK- [6]
Местоположение: Ø64, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 века н.э.
Y-ДНК: I-Y79817
FTDNA Комментарий : Делится 3 SNP с мужчиной из Норвегии ниже I-Y24625.Новая ветвь = I-Y79817
мтДНК: h2ao
Образец: VK190 / Гренландия, поздний-0996
Местоположение: Ø149, Восточное поселение, Гренландия
Возраст: Поздний скандинавский 1360 г. н.э.
Y-ДНК: I-FGC15543
FTDNA Комментарий: Сплит I- FGC15561. Получено 11 предков для 6. Новый путь = I-FGC15543> I-FGC15561
мтДНК: K1a-T195C!
Образец: VK201 / Orkney_Buckquoy, sk M12
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Викинг 5-6 века н.э.
Y-ДНК: I-B293
mtDNA1
Образец: VK202 / Orkney_Buckquoy, sk 7B
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Viking 10 век н.э.
Y-ДНК: R-A151
mtDNA1
Образец: VK203 / Orkney_BY78, Ar.1, sk 3
Расположение: Brough_Road_Birsay, Оркни, Шотландия, Великобритания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY10450
FTDNA Комментарий: FT83323-
0 mtDNA1 9a a h5a
Образец: VK204 / Orkney_Newark for Brothwell
Местоположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY115469
mtDNA 9:
8 h2
Образец: VK205 / Orkney_Newark 68/12
Расположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-YP4345
mtDNA:
8 h4
Образец: VK210 / Poland_Kraków-Zakrzówek gr.24
Местоположение: Краков, Польша
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: I-Z16971
мтДНК: H5e1a1
Образец: VK211 / Польша_Cedynia gr. 435
Местоположение: Cedynia, Poland
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: R-M269
мтДНК: W6
Образец: VK212 / Польша_Cedynia gr. 558
Расположение: Сединия, Польша
Возраст: Викинг 11-12 века н.э.
Y-ДНК: R-CTS11962
мтДНК: h2-T152C!
Образец: VK215 / Дания_Гердруп-Б; sk 1
Местоположение: Гердруп, Силенд, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-M269
мтДНК: J1c2k
Образец: VK217 / Sweden_Ljungbacka
Местоположение: Ljungbacka, Мальме, Швеция
Возраст: Викинг 9-12 вв. Н.э.
Y-ДНК: R-L151
мтДНК: J1b1b1
Образец: VK218 / Russia_Ladoga_5680-4
Расположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: R-BY2848
мтДНК: H5
Образец: VK219 / Russia_Ladoga_5680-10
Местоположение: Ладога, Россия
Возраст: Викинг 10-11 века н.э.
Y-ДНК: I-Y22024
mtDNA: T2b6a
Образец: VK220 / Russia_Ladoga_5680-11
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-FT253975
FTDNA Комментарий: CTS2208 +, BY47171-, CTS7676-, Y20288-, BY69785-, FT253975 +
мтДНК: J2b1a
Образец: VK221 / Russia_Ladoga_5757-14
Местоположение: Ладога, Россия
Возраст: Викинг 9-10 вв. Н.э.
Y-ДНК: I-Y5473
мтДНК: K1d
Образец: VK223 / Россия_Гнездово 75-140
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: I-BY67763
мтДНК: h23a1a1c
Образец: VK224 / Россия_Гнездово 78-249
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: N-CTS2929
мтДНК: H7a1
Образец: VK225 / Iceland_A108
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-BY
мтДНК: h4v-T16093C
Образец: ВК232 / Готланд_Коппарсвик-240.65
Расположение: Коппарсвик, Готланд, Швеция
Возраст: Викинг 900-1050 CE
Y-ДНК: R-Y16505
FTDNA Комментарий: Спекулятивное размещение — U106 +, но U106 (C> T) в древние образцы могут ввести в заблуждение. LAV010, NA34, I7779, ble007, R55 и EDM124 — все это древние образцы, не относящиеся к R и являющиеся U106 +. Более консервативное размещение — R-P310
мтДНК: N1a1a1
Образец: VK234 / Faroe_2
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-FT381000
FTDNA Комментарий: То же разделение, что и VK25.У них общий маркер FT381000 (26352237 T> G)
мтДНК: h4a1a
Образец: VK237 / Faroe_15
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S6355
mtDNA: J2a2c
Образец: VK238 / Faroe_4
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-YP396
mtDNA: h4a1a
Образец: VK239 / Faroe_5
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-M269
mtDNA: H5
Образец: VK242 / Faroe_3
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S764
mtDNA: h4a1a
Образец: VK244 / Faroe_12
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-CTS4179
mtDNA: h3a2a2
Образец: VK245 / Faroe_16
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-BY202785
FTDNA Комментарий: Образует ветвь с VK46 вниз из R-BY202785 (Z287).Новая ветвь = R-FT383000
мтДНК: h4a1
Образец: VK248 / Faroe_22
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: I-M253
мтДНК: h59a
Образец: VK251 / Gotland_Kopparsvik-30.64
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-M459
mtDNA: 9 U5b1e
Образец: VK256 / UK_Dorset-3722
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-YP5718
m
Образец: VK257 / UK_Dorset-3723
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-Y19934
ma
Образец: VK258 / UK_Dorset-3733
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-YP1395
FTDNA Комментарий: FTDNA 5 SNP с мужчиной из Норвегии.Образует новую ветвь R-YP1395. Новая ветвь = R-Ph520
мтДНК: K1a4a1
Образец: VK259 / UK_Dorset-3734
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-FT20255
Оба VK449 и VK259 делят 3 SNP с мужчиной из Швеции. Образует новую ветвь вниз R-FT20255 (Z18). Новая ветвь = R-FT22694
мтДНК: I2
Образец: VK260 / UK_Dorset-3735
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК:
0t Q-BY77336
m Образец: VK261 / UK_Dorset-3736
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-BY64643
Y-ДНК: R-BY64643
m
Образец: VK262 / UK_Dorset-3739
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-FT347811
FTDNA Комментарий: FT347811
2 SNP с американцем неизвестного происхождения.Образует новую ветвь вниз от Y6908 (Z140). В то же время была обнаружена новая ветвь, которая объединяет эту новую древнюю / американскую ветвь с установленной веткой I-FT274828. Новый древний путь = I-Y6908> I-FT273257> I-FT347811
мтДНК: J1c4
Образец: VK263 / UK_Dorset-3742
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: K-Z16372
m
Образец: VK264 / UK_Dorset-3744
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Образец: VK267 / Sweden_Karda 21
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-L23
мтДНК: T2b4b
Образец: VK268 / Sweden_Karda 22
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
мтДНК: K1c1
Образец: VK269 / Sweden_Karda 24
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
mtDNA: h2e1a
Образец: VK273 / Россия_Гнездово 77-255
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: R-BY61747
мтДНК: U5a2a1b1
Образец: VK274 / Дания_Kaargarden 391
Местоположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-Ph4519
mtDNA: T2b-T152C!
Образец: VK275 / Дания_Kaargarden 217
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY74743
мтДНК: H
Образец: VK279 / Дания_Galgedil AX
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y10639
мтДНК: I4a
Образец: VK280 / Дания_Galgedil UO
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y3713
мтДНК: h21a
Образец: VK281 / Дания_Barse Grav A
Местоположение: Борсе, Силенд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-FGC22153
Комментарий FTDNA: Разделяет I-Y5612 ( P109).Получено для 8, предков для 2. Новый путь = I-Y5612> I-Y5619
мтДНК: T2
Образец: VK282 / Дания_Stengade I, LMR c195
Местоположение: Stengade_I, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-CTS1211
mtDNA: 8 h5a
Образец: VK286 / Дания_Bogovej Grav BJ
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-S10708
mtDNA: J1c-C16261
Образец: VK287 / Дания_Kaargarden Grav BS
Местоположение: Kaagården, Langeland, Дания
Возраст: Viking X век н.э.
Y-ДНК: R-S22676
mtDNA: T2b
Образец: VK289 / Дания_Bodkergarden Grav H, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-U106
mtDNA:
8 J2b1a
Образец: VK290 / Дания_Kumle Hoje Grav O
Местоположение: Kumle_høje, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-FT264183
FTDNA Комментарий: Акционирует не менее SNP с мужчиной из Швеции, образуя новую ветвь ниже по течению R-FT263905 (U106).Новая ветка = R-FT264183. HG02545 остается в R-FT263905
мтДНК: I1a1
Образец: VK291 / Дания_Bodkergarden Grav D, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг, 9 век н.э.
Y-ДНК: I-Y20861
mtDNA: U5a
Образец: VK292 / Дания_Bogovej Grav A.D.
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-M417
мтДНК:
028 J1c2c1 9 Образец: VK295 / Дания_Hessum sk 1
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y4738
мтДНК: T1a1
Образец: VK296 / Дания_Hundstrup Mose sk 1
Расположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 660-780 CE
Y-ДНК: I-S7660
mtDNA: HV6
Образец: VK297 / Дания_Hundstrup Mose sk 2
Местоположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 670-830 CE
Y-ДНК: I-Y4051
mtDNA:
0 J1c2h 9 Образец: VK301 / Дания_Ladby Grav 4
Местоположение: Ladby, Funen, Дания
Возраст: Viking 640-890 CE
Y-ДНК: I-FT105192
mtDNA: R0a2b
Образец: VK306 / Sweden_Skara 33
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: I-FT115400
FTDNA Комментарий: Имеет 3 мутации с мужчина из Швеции.Образует новую ветвь I-S19291. Новая ветка = I-FT115400. VK151 не имеет покрытия для 2 из этих мутаций
мтДНК: h25a1
Образец: VK308 / Sweden_Skara 101
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: R-BY33037
мтДНК: h2c
Образец: VK309 / Sweden_Skara 53
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-YP6189
мтДНК: K1b1c
Образец: VK313 / Дания_Rantzausminde Grav 2
Местоположение: Ранцаусминде, Фюн, Дания
Возраст: Викинг 850-900 CE
Y-ДНК: R-JFS0009
мтДНК: h2b
Образец: VK315 / Дания_Bakkendrup Grav 16
Местоположение: Баккендруп, Силенд, Дания
Возраст: Viking 850-900 CE
Y-ДНК: I-Y98280
Комментарий FTDNA: Делится 1 SNP с мужчина из Нидерландов.Образует новую ветвь после I-Y37415 (P109). Новая ветвь = I-Y98280
мтДНК: T1a1b
Образец: VK316 / Дания_Hessum sk II
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y130659
Комментарий FTDNA: Сплит I- Y130594 (Z59). Получено для 1 предка для 6. Новый путь = I-Y130659> I-Y130594> I-Y130747. Древний образец STR_486 также принадлежит к этой группе, по I-Y130747
мтДНК: K1a4
Образец: VK317 / Дания_Kaargarden Grav BF99
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: J-BY62479
FTDNA Комментарий: 6-Splits J2 M67).Получено для 9, предков для 3. Новый путь = J-BY62479> J-BY72550
мтДНК: h3a2a1
Образец: VK320 / Дания_Bogovej Grav S
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y103013
Комментарий FTDNA: Делится 3 SNP с мужчина из Швеции. Образует новую ветвь вниз I-FT3562 (P109). Новая ветвь = I-Y103013
мтДНК: U5a1a1
Образец: VK323 / Дания_Рибе 2
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-S10185
мтДНК: K2a6
Образец: VK324 / Дания_Ribe 3
Расположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY16590
FTDNA Комментарий: Сплит R-BY16590 (L47).Получено для 7, предков для 3. Новый путь = R-S9742> R-BY16950
мтДНК: N1a1a1a2
Образец: VK326 / Дания_Рибе 5
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-Y52895
mtDNA: U5b1-T16189C! — T16192C!
Образец: VK327 / Дания_Рибе 6
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-BY463
мтДНК: H6a1a5
Образец: VK329 / Дания_Ribe 8
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинги 9-11 веков н.э.
Y-ДНК: R-S18894
мтДНК: h4-T152C!
Образец: VK332 / Oland_1088
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-S8522
FTDNA Комментарий: Возможно, ниже I-BY195155.Имеет одну мутацию C> T с образцом BY195155 *
мтДНК: T2b24
Образец: VK333 / Oland_1028
Местоположение: Эланд, Швеция
Возраст: Viking 885 ± 69 CE
Y-ДНК: R-Z29034
mtDNA: h3a2a1
Образец: VK335 / Oland_1068
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н. Э.
Y-ДНК: R-BY39347
FTDNA Комментарий: Делится 8 SNP с мужчиной Франция.Образует новую ветвь вниз по R-BY39347 (U152). Новая ветвь = R-FT304388
мтДНК: K1b2a3
Образец: VK336 / Oland_1075
Местоположение: Эланд, Швеция
Возраст: Viking 853 ± 67 CE
Y-ДНК: R-BY106906
мтДНК: K2a3a
Образец: VK337 / Oland_1064
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-BY31739
FTDNA Комментарий: Возможный Z140
mtDNA1b: 9003a U5a
Образец: VK338 / Дания_Bogovej Grav BV
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-A6707
мтДНК: W3a1
Образец: VK342 / Oland_1016
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-BY78615
FTDNA Комментарий: Имеет 2 SNP с мужчиной из Финляндия.Образует новую ветвь вниз I2-Y23710 (L801). Новая ветвь = I-BY78615
мтДНК: h3a1
Образец: VK343 / Oland_1021
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y7232
мтДНК: h4h
Образец: VK344 / Oland_1030
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32357
mtDNA: J1c2t
Образец: VK345 / Oland_1045
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-FT148754
FTDNA Комментарий: Сплит R-FT148754 (DF63) .Получено для 8, предков для 6. Новый путь = R-FT148796> R-FT148754
мтДНК: h5a1
Образец: VK346 / Oland_1057
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 века н.э.
Y-ДНК: J-Z8424
мтДНК: h3a2b
Образец: VK348 / Oland_1067
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Z171
мтДНК: T2b28
Образец: VK349 / Oland_1073
Местоположение: Оланд, Швеция
Возраст: Viking 829 ± 57 CE
Y-ДНК: R-BY166065
FTDNA Комментарий: Делится 2 SNP с мужчиной из Англии .Образует ответвление вниз от R-BY166065 (L1066). Новая ветвь = R-BY167052
мтДНК: h2e2a
Образец: VK352 / Oland_1012
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-FGC35755
FTDNA Комментарий: Возможно, образует ветвь вниз I -Y15295. 2 возможные мутации G> A с образцом I-Y15295 *
мтДНК: H64
Образец: VK354 / Oland_1026
Местоположение: Эланд, Швеция
Возраст: Viking 986 ± 38 CE
Y-ДНК: R-S6752
мтДНК: h3a1
Образец: VK355 / Oland_1046
Местоположение: Эланд, Швеция
Возраст: Викинг 847 ± 65 CE
Y-ДНК: L-L595
FTDNA Комментарий: Присоединяется к 2 другим древним на этой редкой ветви .ASH087 и I2923
мтДНК: U5b1b1a
Образец: VK357 / Oland_1097
Местоположение: Эланд, Швеция
Возраст: Viking 1053 ± 60 CE
Y-ДНК: I-FT49567
FTDNA Комментарий: Делится 4 SNP с мужчиной из Англии . Образует новую ветвь I-A5952 (Z140). Новая ветвь = I-FT49567
мтДНК: J2b1a
Образец: VK362 / Дания_Bogovej LMR 12077
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: E-CTS5856
FTDNA Комментарий:
3 Возможно 90 E-Z мтДНК: V7b
Образец: VK363 / Дания_Bogovej BT
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY198083
Комментарий FTDNA: Делится 2 SNP с мужчиной из Швейцарии.Образует новую ветвь I-A1472 (Z140). Новая ветвь = I-BY198083
мтДНК: U4b1a1a1
Образец: VK365 / Дания_Bogovej BS
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY34800
мтДНК: U8a2
Образец: VK367 / Дания_Bogovej D
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY67827
Комментарий FTDNA: VK506 и VK19967 9 Внутренние отделы — SCP Foundation
Успешная работа фонда зависит, прежде всего, от разделения ответственности между различными отделами.Некоторые подразделения Фонда перечислены в этом документе. Ради эффективности все отделы тесно координируются.
Административный отдел
Административный отдел возглавляют непосредственно команда O5 и администратор. Он направляет действия Фонда на всех уровнях. В этот отдел входят все узлы связи и командные центры; у каждого такого узла и центра есть резерв на случай отказа.В юрисдикцию AD также входят финансовые операции Фонда в целом, такие как распоряжение средствами, бухгалтерский учет, планирование бюджета и т. Д. Из-за важности этого отдела проверки лояльности здесь проводятся чаще, чем где-либо еще. К этому же отделу относится делопроизводство. Департамент иностранных дел (DEA)
DEA отвечает за широкий спектр операций: распространение дезинформации, устранение следов деятельности SCP (как объектов, так и деятельности Фонда), набор персонала класса D и набор новых сотрудников из военных и гражданских учреждений.DEA отвечает за сокрытие деятельности агентов и объектов Фонда в населенных пунктах. DEA — это, по сути, передовая линия Фонда, поскольку она фильтрует всех потенциальных сотрудников и отвечает за поддержание высоких стандартов среди нанятого персонала — это также можно назвать HR. Отдел инженерно-технического обслуживания
E&TS отвечает за техническое обслуживание и повседневное обслуживание всех объектов и оборудования, активно используемых Фондом.Этот отдел отвечает за проектирование и строительство новых объектов и путей сообщения, а также за ремонт и техническое обслуживание. Отдел E&TS разделен на несколько подотделов в зависимости от уровней допуска и ассигнований; сотрудники с более высоким уровнем допуска проектируют и строят камеры содержания класса Кетер и работают со стратегически важными объектами. Комитет по этике
Комитет по этике — небольшой независимый отдел, отвечающий за рассмотрение процедур сдерживания и проверку проведенных экспериментов на предмет чрезмерного расточительства или ненадлежащего использования сотрудников класса D и других ресурсов.Некоторые считают существование комитета плохой шуткой, но он играет значительную роль в деятельности Фонда и имеет большое влияние. Разведывательное управление
Разведывательное управление занимается поиском, отслеживанием и захватом несохраняемых объектов SCP, а также сбором разведданных о враждебных группах интересов. Ячейки под прикрытием IA существуют во всех крупных населенных пунктах и замаскированы под гражданские объекты и учреждения.Агенты разведки обычно набираются из различных силовых структур и спецслужб мира. Отдел внутренней безопасности
ISD — это скрытый «фонд в Фонде», силы тайной полиции, ответственные за фильтрацию предателей, а также за оперативные риски и риски информационной безопасности в рядах Фонда. Отдел строго иерархичен, придерживаясь строго определенного многоэтапного протокола расследования.Потенциальные агенты ISD должны иметь безупречный стаж работы и стаж работы в Фонде более года. Обычно каждый агент ISD сохраняет свою «официальную» позицию, чтобы предоставить своей команде отчет из первых рук обо всем, что происходит в данном отделе. Само существование ISD обычно преподносится как миф. «Официальная» ответственность ИСД — это расследование и допрос захваченных агентов ГОИ, хотя эта часть ИСД официально принадлежит Управлению безопасности в целях секретности. Отдел логистики
Департамент логистики отвечает за передачу природных, финансовых и человеческих ресурсов между объектами Фонда. Транспортировка осуществляется автомобильным, водным, железнодорожным, воздушным, космическим и другим транспортом. Имеется сложная система распределения приоритетов и классов важности перевозок; Объекты SCP и ресурсы, необходимые для их сдерживания, обычно получают высший приоритет.Устранение препятствий, возникающих при транспортировке, также является первоочередной задачей. Производственный отдел
Производственный отдел отвечает за выполнение любых материальных запросов, которые могут возникнуть во время поимки, содержания или любого другого процесса Фонда. Производственный отдел имеет несколько участков и зон, полностью посвященных созданию широкого профиля изделий на заказ или серийного производства. Проектирование и производство часто осуществляется на месте по соображениям безопасности.По тем же причинам ни производственный персонал, ни проектная документация никогда не покидают пределы объекта. Обычно они оснащены современным оборудованием и тщательно охраняются. Лечебное отделение
Медицинский отдел Фонда отвечает за поддержание хорошего здоровья сотрудников. Из-за характера работы Фонда медики часто сталкиваются с трудно поддающимися лечению и весьма необычными случаями ран, травм, инфекций и расстройств.В связи с этим ОД придерживается исключительно высоких кадровых стандартов. Некоторые медики Фонда специализируются на вторичных областях лечения, таких как меметические воздействия, психологические травмы и т. Д. Мобильные оперативные группы
Работа Фонда требует большого количества узкоспециализированных специалистов. Обычно это связано с аномалиями, которые требуют больших усилий и нестандартного подхода к локализации.Для решения этой проблемы создаются мобильные оперативные группы. MTF — это группы разного размера, которые специализируются на определенных ролях или реагируют на конкретные ситуации. У каждой группы есть название, состоящее из греческой буквы и числа; уточняющие префиксы и / или позывные могут использоваться в некоторых случаях. Дополнительную информацию можно найти в Обзоре мобильных оперативных групп. Научный отдел
Эффективная научная работа — краеугольный камень существования и деятельности Фонда.Научный отдел отвечает за изучение всех содержащихся и новых SCP-объектов, разработку мер противодействия событиям нарушения условий содержания и разработку различных устройств и лекарственных препаратов, которые будут производиться Производственным отделом. Сотрудники научного отдела набираются из числа лучших и перспективных ученых в различных областях. Отдел безопасности
Этот отдел отвечает за обеспечение безопасности и защиты всех помещений и объектов Фонда, независимо от их приоритета.Сотрудники SD обучены противодействию событиям нарушения условий содержания, вторжениям, саботажу, стихийным бедствиям и другим чрезвычайным ситуациям. Новички в этой услуге обычно проходят общее обучение перед тем, как попасть на объект или площадку. Специальное обучение работе с данным объектом проводится на выезде после задания. Департамент внутреннего трибунала
Отдел внутреннего трибунала Фонда SCP является институциональным судебным органом организации.Этот отдел, по просьбе ISD, рассматривает дела сотрудников Фонда и других лиц, которые подпадают под определение запрещенных Правовым кодексом Департамента внутреннего трибунала, включая такие записи, как: «I — Преступления против Фонда», «II — Преступления против нормальности и человечности »,« III — Существенные дисциплинарные нарушения »,« IV — Подготовка к проведению мероприятия класса K »,« V — Преступление государства и корпорации »,« VI — Прочие нарушения ». Трибунал может назначить различные приговоры по каждой категории нарушений (за исключением пятого, судебное разбирательство по которому ведется ГОК).Любое решение Трибунала может быть отменено Командованием O5. Вы можете ознакомиться с Положением о внутреннем трибунале Фонда SCP и его Юридическим кодексом здесь. Центральные базы данных мутаций и SNP
Репозиторий dbSNP-полиморфизмов NCBI, NIH, Bethesda Сервер молекулярной биологии ExPASy Швейцарский институт биоинформатики (SIB) Европейский институт биоинформатики EBI EMBL / EBI GeneSNPs (общедоступный Интернет-ресурс) Команда Центра генома Университета Юты ClinVar, NCBI Национальный центр биотехнологической информации Генатлас Жан Фрезаль, INSERM, Генетон, Франция, База данных генных карт М.Ребхан, Дж. Прилуски, В. Чалифа-Каспи и Дорон Ланцет Институт Вейцмана, Реховот, Израиль GVS: сервер изменения генома Д-р Дебора Никерсон, д-р Марк Ридер и команда США HGVbase: база данных вариаций генома человека Каролинский институт, Стокгольм, Швеция Европейский институт биоинформатики (EBI), Хинкстон, Великобритания и Европа, лаборатория молекулярной биологии (EMBL), Гейдельберг, Германия HOWDY: Организованная база данных полного генома человека Японская научно-техническая корпорация База данных мутаций генов человека, HGMD Д.Н. Купер, Э. В. Болл, П. Д. Стенсон М. Кравчак, Univ. Медицинского колледжа Уэльса. Кардифф, Великобритания jSNP: база База данных японских SNP Центр генома человека, Институт медицинских наук, Токийский университет База данных SNP Leelab Иризарри, К. Кустанович, В. Ли, К.Браун, Н. Нельсон, С. Вонг, В., Ли, К.Дж. UCLA Центр биоинформатики База данных человеческих SNP Институт Уайтхеда / Центр исследований генома Массачусетского технологического института Интернет-Менделирующее наследование в человеке, OMIM В. А. МакКусик и его коллеги, Университет Джона Хопкинса, NCBI NIEHS SNP ДокторДебора Никерсон, доктор Марк Ридер и команда США PharmGKB: База знаний по фармакогенетике и фармакогеномике Стэнфордский университет США SNP в Сиэтле Д-р Дебора Никерсон, д-р Марк Ридер и их команда Вашингтонского университета, Принстонского университета и Национального института сердца, легких и крови, U.
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT7019
mtDNA1:
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT13004
FTDNA Комментарий: Расщепляет I2-FT12648, полученный для 5, наследственный для 7.Новый путь FT13004> FT12648
мтДНК: U5a1b-T16362C
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FGC17429
mtDNA -C16239T
Местоположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-Y47841
FTDNA Комментарий: Делит 6 SNP с человеком из Швеции, ниже R-BY38950 (R-Y47841)
мтДНК: h2a1
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: I-FT3562
mtDNA: mtDNA:
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-FT31867
FTDNA Комментарий: Делится 3 SNP с мужчиной из Греции.Образует новую ветвь после R-BY220332 (U152). Новая ветвь = R-FT31867
мтДНК: H82
Расположение: St_John’s_College_Oxford, Oxford, England, UK
Возраст: Viking 880-1000 CE
Y-ДНК: R-BY176639
FTDNA Комментарий: Соединяется с PGA3 (Проект персонального генома, Австрия) и клиентом FTDNA из Дании. Клиенты PGA и FTDNA сформировали филиал ранее на этой неделе, VK178 присоединится к ним по адресу R-BY176639 (под L48)
мтДНК: K2a5
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Раннее скандинавское население 10–12 веков н. Э.
Y-ДНК: I-F3312
мтДНК: K1a3a
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н.э.
Y-ДНК: I-F3312
мтДНК: T2b21
Расположение: Ø029a, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 вв. Н. Э.
Y-ДНК: R-YP4342
мтДНК: h5a1a4b
Местоположение: Ø64, Восточное поселение, Гренландия
Возраст: Ранние скандинавские 10-12 века н.э.
Y-ДНК: I-Y79817
FTDNA Комментарий : Делится 3 SNP с мужчиной из Норвегии ниже I-Y24625.Новая ветвь = I-Y79817
мтДНК: h2ao
Местоположение: Ø149, Восточное поселение, Гренландия
Возраст: Поздний скандинавский 1360 г. н.э.
Y-ДНК: I-FGC15543
FTDNA Комментарий: Сплит I- FGC15561. Получено 11 предков для 6. Новый путь = I-FGC15543> I-FGC15561
мтДНК: K1a-T195C!
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Викинг 5-6 века н.э.
Y-ДНК: I-B293
mtDNA1
Местоположение: Buckquoy_Birsay, Orkney, Scotland, UK
Возраст: Viking 10 век н.э.
Y-ДНК: R-A151
mtDNA1
Расположение: Brough_Road_Birsay, Оркни, Шотландия, Великобритания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY10450
FTDNA Комментарий: FT83323-
Местоположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY115469
mtDNA 9:
Расположение: Newark_Deerness, Orkney, Scotland, UK
Возраст: Викинг 10 век н.э.
Y-ДНК: R-YP4345
mtDNA:
Местоположение: Краков, Польша
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: I-Z16971
мтДНК: H5e1a1
Местоположение: Cedynia, Poland
Возраст: Средневековье 11-13 вв. Н. Э.
Y-ДНК: R-M269
мтДНК: W6
Расположение: Сединия, Польша
Возраст: Викинг 11-12 века н.э.
Y-ДНК: R-CTS11962
мтДНК: h2-T152C!
Местоположение: Гердруп, Силенд, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-M269
мтДНК: J1c2k
Местоположение: Ljungbacka, Мальме, Швеция
Возраст: Викинг 9-12 вв. Н.э.
Y-ДНК: R-L151
мтДНК: J1b1b1
Расположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: R-BY2848
мтДНК: H5
Местоположение: Ладога, Россия
Возраст: Викинг 10-11 века н.э.
Y-ДНК: I-Y22024
mtDNA: T2b6a
Местоположение: Ладога, Россия
Возраст: Викинг 10-12 вв. Н.э.
Y-ДНК: I-FT253975
FTDNA Комментарий: CTS2208 +, BY47171-, CTS7676-, Y20288-, BY69785-, FT253975 +
мтДНК: J2b1a
Местоположение: Ладога, Россия
Возраст: Викинг 9-10 вв. Н.э.
Y-ДНК: I-Y5473
мтДНК: K1d
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: I-BY67763
мтДНК: h23a1a1c
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: N-CTS2929
мтДНК: H7a1
Расположение: Хофстадир, Исландия
Возраст: Викинг 10-13 вв. Н. Э.
Y-ДНК: R-BY
мтДНК: h4v-T16093C
Расположение: Коппарсвик, Готланд, Швеция
Возраст: Викинг 900-1050 CE
Y-ДНК: R-Y16505
FTDNA Комментарий: Спекулятивное размещение — U106 +, но U106 (C> T) в древние образцы могут ввести в заблуждение. LAV010, NA34, I7779, ble007, R55 и EDM124 — все это древние образцы, не относящиеся к R и являющиеся U106 +. Более консервативное размещение — R-P310
мтДНК: N1a1a1
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-FT381000
FTDNA Комментарий: То же разделение, что и VK25.У них общий маркер FT381000 (26352237 T> G)
мтДНК: h4a1a
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S6355
mtDNA: J2a2c
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-YP396
mtDNA: h4a1a
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-M269
mtDNA: H5
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-S764
mtDNA: h4a1a
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-CTS4179
mtDNA: h3a2a2
Расположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: R-BY202785
FTDNA Комментарий: Образует ветвь с VK46 вниз из R-BY202785 (Z287).Новая ветвь = R-FT383000
мтДНК: h4a1
Местоположение: Church3, Фарерские острова
Возраст: Раннее Новое время 16-17 веков н.э.
Y-ДНК: I-M253
мтДНК: h59a
Местоположение: Коппарсвик, Готланд, Швеция
Возраст: Viking 900-1050 CE
Y-ДНК: R-M459
mtDNA: 9 U5b1e
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-YP5718
m
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-Y19934
ma
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-YP1395
FTDNA Комментарий: FTDNA 5 SNP с мужчиной из Норвегии.Образует новую ветвь R-YP1395. Новая ветвь = R-Ph520
мтДНК: K1a4a1
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК: R-FT20255
Оба VK449 и VK259 делят 3 SNP с мужчиной из Швеции. Образует новую ветвь вниз R-FT20255 (Z18). Новая ветвь = R-FT22694
мтДНК: I2
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Dorset, England, UK
Возраст: Viking 10-11 веков н.э.
Y-ДНК:
m
Образец: VK261 / UK_Dorset-3736
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: R-BY64643
Y-ДНК: R-BY64643
m
Образец: VK262 / UK_Dorset-3739
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: I-FT347811
FTDNA Комментарий: FT347811
2 SNP с американцем неизвестного происхождения.Образует новую ветвь вниз от Y6908 (Z140). В то же время была обнаружена новая ветвь, которая объединяет эту новую древнюю / американскую ветвь с установленной веткой I-FT274828. Новый древний путь = I-Y6908> I-FT273257> I-FT347811
мтДНК: J1c4
Образец: VK263 / UK_Dorset-3742
Местоположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Y-ДНК: K-Z16372
m
Образец: VK264 / UK_Dorset-3744
Расположение: Ridgeway_Hill_Mass_Grave_Dorset, Дорсет, Англия, Великобритания
Возраст: Викинг 10-11 веков н.э.
Образец: VK267 / Sweden_Karda 21
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-L23
мтДНК: T2b4b
Образец: VK268 / Sweden_Karda 22
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
мтДНК: K1c1
Образец: VK269 / Sweden_Karda 24
Местоположение: Карда, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-M269
mtDNA: h2e1a
Образец: VK273 / Россия_Гнездово 77-255
Местоположение: Гнездово, Россия
Возраст: Викинг 10-11 вв. Н.э.
Y-ДНК: R-BY61747
мтДНК: U5a2a1b1
Образец: VK274 / Дания_Kaargarden 391
Местоположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-Ph4519
mtDNA: T2b-T152C!
Образец: VK275 / Дания_Kaargarden 217
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY74743
мтДНК: H
Образец: VK279 / Дания_Galgedil AX
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y10639
мтДНК: I4a
Образец: VK280 / Дания_Galgedil UO
Местоположение: Галгедил, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y3713
мтДНК: h21a
Образец: VK281 / Дания_Barse Grav A
Местоположение: Борсе, Силенд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-FGC22153
Комментарий FTDNA: Разделяет I-Y5612 ( P109).Получено для 8, предков для 2. Новый путь = I-Y5612> I-Y5619
мтДНК: T2
Образец: VK282 / Дания_Stengade I, LMR c195
Местоположение: Stengade_I, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-CTS1211
mtDNA: 8 h5a
Образец: VK286 / Дания_Bogovej Grav BJ
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-S10708
mtDNA: J1c-C16261
Образец: VK287 / Дания_Kaargarden Grav BS
Местоположение: Kaagården, Langeland, Дания
Возраст: Viking X век н.э.
Y-ДНК: R-S22676
mtDNA: T2b
Образец: VK289 / Дания_Bodkergarden Grav H, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг 9 век н.э.
Y-ДНК: R-U106
mtDNA:
8 J2b1a
Образец: VK290 / Дания_Kumle Hoje Grav O
Местоположение: Kumle_høje, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-FT264183
FTDNA Комментарий: Акционирует не менее SNP с мужчиной из Швеции, образуя новую ветвь ниже по течению R-FT263905 (U106).Новая ветка = R-FT264183. HG02545 остается в R-FT263905
мтДНК: I1a1
Образец: VK291 / Дания_Bodkergarden Grav D, sk 1
Местоположение: Bødkergarden, Langeland, Дания
Возраст: Викинг, 9 век н.э.
Y-ДНК: I-Y20861
mtDNA: U5a
Образец: VK292 / Дания_Bogovej Grav A.D.
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-M417
мтДНК:
028 J1c2c1 9 Образец: VK295 / Дания_Hessum sk 1
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y4738
мтДНК: T1a1
Образец: VK296 / Дания_Hundstrup Mose sk 1
Расположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 660-780 CE
Y-ДНК: I-S7660
mtDNA: HV6
Образец: VK297 / Дания_Hundstrup Mose sk 2
Местоположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 670-830 CE
Y-ДНК: I-Y4051
mtDNA:
0 J1c2h 9 Образец: VK301 / Дания_Ladby Grav 4
Местоположение: Ladby, Funen, Дания
Возраст: Viking 640-890 CE
Y-ДНК: I-FT105192
mtDNA: R0a2b
Образец: VK306 / Sweden_Skara 33
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: I-FT115400
FTDNA Комментарий: Имеет 3 мутации с мужчина из Швеции.Образует новую ветвь I-S19291. Новая ветка = I-FT115400. VK151 не имеет покрытия для 2 из этих мутаций
мтДНК: h25a1
Образец: VK308 / Sweden_Skara 101
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: R-BY33037
мтДНК: h2c
Образец: VK309 / Sweden_Skara 53
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-YP6189
мтДНК: K1b1c
Образец: VK313 / Дания_Rantzausminde Grav 2
Местоположение: Ранцаусминде, Фюн, Дания
Возраст: Викинг 850-900 CE
Y-ДНК: R-JFS0009
мтДНК: h2b
Образец: VK315 / Дания_Bakkendrup Grav 16
Местоположение: Баккендруп, Силенд, Дания
Возраст: Viking 850-900 CE
Y-ДНК: I-Y98280
Комментарий FTDNA: Делится 1 SNP с мужчина из Нидерландов.Образует новую ветвь после I-Y37415 (P109). Новая ветвь = I-Y98280
мтДНК: T1a1b
Образец: VK316 / Дания_Hessum sk II
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y130659
Комментарий FTDNA: Сплит I- Y130594 (Z59). Получено для 1 предка для 6. Новый путь = I-Y130659> I-Y130594> I-Y130747. Древний образец STR_486 также принадлежит к этой группе, по I-Y130747
мтДНК: K1a4
Образец: VK317 / Дания_Kaargarden Grav BF99
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: J-BY62479
FTDNA Комментарий: 6-Splits J2 M67).Получено для 9, предков для 3. Новый путь = J-BY62479> J-BY72550
мтДНК: h3a2a1
Образец: VK320 / Дания_Bogovej Grav S
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y103013
Комментарий FTDNA: Делится 3 SNP с мужчина из Швеции. Образует новую ветвь вниз I-FT3562 (P109). Новая ветвь = I-Y103013
мтДНК: U5a1a1
Образец: VK323 / Дания_Рибе 2
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-S10185
мтДНК: K2a6
Образец: VK324 / Дания_Ribe 3
Расположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY16590
FTDNA Комментарий: Сплит R-BY16590 (L47).Получено для 7, предков для 3. Новый путь = R-S9742> R-BY16950
мтДНК: N1a1a1a2
Образец: VK326 / Дания_Рибе 5
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-Y52895
mtDNA: U5b1-T16189C! — T16192C!
Образец: VK327 / Дания_Рибе 6
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-BY463
мтДНК: H6a1a5
Образец: VK329 / Дания_Ribe 8
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинги 9-11 веков н.э.
Y-ДНК: R-S18894
мтДНК: h4-T152C!
Образец: VK332 / Oland_1088
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-S8522
FTDNA Комментарий: Возможно, ниже I-BY195155.Имеет одну мутацию C> T с образцом BY195155 *
мтДНК: T2b24
Образец: VK333 / Oland_1028
Местоположение: Эланд, Швеция
Возраст: Viking 885 ± 69 CE
Y-ДНК: R-Z29034
mtDNA: h3a2a1
Образец: VK335 / Oland_1068
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н. Э.
Y-ДНК: R-BY39347
FTDNA Комментарий: Делится 8 SNP с мужчиной Франция.Образует новую ветвь вниз по R-BY39347 (U152). Новая ветвь = R-FT304388
мтДНК: K1b2a3
Образец: VK336 / Oland_1075
Местоположение: Эланд, Швеция
Возраст: Viking 853 ± 67 CE
Y-ДНК: R-BY106906
мтДНК: K2a3a
Образец: VK337 / Oland_1064
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-BY31739
FTDNA Комментарий: Возможный Z140
mtDNA1b: 9003a U5a
Образец: VK338 / Дания_Bogovej Grav BV
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-A6707
мтДНК: W3a1
Образец: VK342 / Oland_1016
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-BY78615
FTDNA Комментарий: Имеет 2 SNP с мужчиной из Финляндия.Образует новую ветвь вниз I2-Y23710 (L801). Новая ветвь = I-BY78615
мтДНК: h3a1
Образец: VK343 / Oland_1021
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y7232
мтДНК: h4h
Образец: VK344 / Oland_1030
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32357
mtDNA: J1c2t
Образец: VK345 / Oland_1045
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-FT148754
FTDNA Комментарий: Сплит R-FT148754 (DF63) .Получено для 8, предков для 6. Новый путь = R-FT148796> R-FT148754
мтДНК: h5a1
Образец: VK346 / Oland_1057
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 века н.э.
Y-ДНК: J-Z8424
мтДНК: h3a2b
Образец: VK348 / Oland_1067
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Z171
мтДНК: T2b28
Образец: VK349 / Oland_1073
Местоположение: Оланд, Швеция
Возраст: Viking 829 ± 57 CE
Y-ДНК: R-BY166065
FTDNA Комментарий: Делится 2 SNP с мужчиной из Англии .Образует ответвление вниз от R-BY166065 (L1066). Новая ветвь = R-BY167052
мтДНК: h2e2a
Образец: VK352 / Oland_1012
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-FGC35755
FTDNA Комментарий: Возможно, образует ветвь вниз I -Y15295. 2 возможные мутации G> A с образцом I-Y15295 *
мтДНК: H64
Образец: VK354 / Oland_1026
Местоположение: Эланд, Швеция
Возраст: Viking 986 ± 38 CE
Y-ДНК: R-S6752
мтДНК: h3a1
Образец: VK355 / Oland_1046
Местоположение: Эланд, Швеция
Возраст: Викинг 847 ± 65 CE
Y-ДНК: L-L595
FTDNA Комментарий: Присоединяется к 2 другим древним на этой редкой ветви .ASH087 и I2923
мтДНК: U5b1b1a
Образец: VK357 / Oland_1097
Местоположение: Эланд, Швеция
Возраст: Viking 1053 ± 60 CE
Y-ДНК: I-FT49567
FTDNA Комментарий: Делится 4 SNP с мужчиной из Англии . Образует новую ветвь I-A5952 (Z140). Новая ветвь = I-FT49567
мтДНК: J2b1a
Образец: VK362 / Дания_Bogovej LMR 12077
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: E-CTS5856
FTDNA Комментарий:
3 Возможно 90 E-Z мтДНК: V7b
Образец: VK363 / Дания_Bogovej BT
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY198083
Комментарий FTDNA: Делится 2 SNP с мужчиной из Швейцарии.Образует новую ветвь I-A1472 (Z140). Новая ветвь = I-BY198083
мтДНК: U4b1a1a1
Образец: VK365 / Дания_Bogovej BS
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY34800
мтДНК: U8a2
Образец: VK367 / Дания_Bogovej D
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY67827
Комментарий FTDNA: VK506 и VK19967 9 Внутренние отделы — SCP Foundation
Успешная работа фонда зависит, прежде всего, от разделения ответственности между различными отделами.Некоторые подразделения Фонда перечислены в этом документе. Ради эффективности все отделы тесно координируются.
Административный отдел
Административный отдел возглавляют непосредственно команда O5 и администратор. Он направляет действия Фонда на всех уровнях. В этот отдел входят все узлы связи и командные центры; у каждого такого узла и центра есть резерв на случай отказа.В юрисдикцию AD также входят финансовые операции Фонда в целом, такие как распоряжение средствами, бухгалтерский учет, планирование бюджета и т. Д. Из-за важности этого отдела проверки лояльности здесь проводятся чаще, чем где-либо еще. К этому же отделу относится делопроизводство. Департамент иностранных дел (DEA)
DEA отвечает за широкий спектр операций: распространение дезинформации, устранение следов деятельности SCP (как объектов, так и деятельности Фонда), набор персонала класса D и набор новых сотрудников из военных и гражданских учреждений.DEA отвечает за сокрытие деятельности агентов и объектов Фонда в населенных пунктах. DEA — это, по сути, передовая линия Фонда, поскольку она фильтрует всех потенциальных сотрудников и отвечает за поддержание высоких стандартов среди нанятого персонала — это также можно назвать HR. Отдел инженерно-технического обслуживания
E&TS отвечает за техническое обслуживание и повседневное обслуживание всех объектов и оборудования, активно используемых Фондом.Этот отдел отвечает за проектирование и строительство новых объектов и путей сообщения, а также за ремонт и техническое обслуживание. Отдел E&TS разделен на несколько подотделов в зависимости от уровней допуска и ассигнований; сотрудники с более высоким уровнем допуска проектируют и строят камеры содержания класса Кетер и работают со стратегически важными объектами. Комитет по этике
Комитет по этике — небольшой независимый отдел, отвечающий за рассмотрение процедур сдерживания и проверку проведенных экспериментов на предмет чрезмерного расточительства или ненадлежащего использования сотрудников класса D и других ресурсов.Некоторые считают существование комитета плохой шуткой, но он играет значительную роль в деятельности Фонда и имеет большое влияние. Разведывательное управление
Разведывательное управление занимается поиском, отслеживанием и захватом несохраняемых объектов SCP, а также сбором разведданных о враждебных группах интересов. Ячейки под прикрытием IA существуют во всех крупных населенных пунктах и замаскированы под гражданские объекты и учреждения.Агенты разведки обычно набираются из различных силовых структур и спецслужб мира. Отдел внутренней безопасности
ISD — это скрытый «фонд в Фонде», силы тайной полиции, ответственные за фильтрацию предателей, а также за оперативные риски и риски информационной безопасности в рядах Фонда. Отдел строго иерархичен, придерживаясь строго определенного многоэтапного протокола расследования.Потенциальные агенты ISD должны иметь безупречный стаж работы и стаж работы в Фонде более года. Обычно каждый агент ISD сохраняет свою «официальную» позицию, чтобы предоставить своей команде отчет из первых рук обо всем, что происходит в данном отделе. Само существование ISD обычно преподносится как миф. «Официальная» ответственность ИСД — это расследование и допрос захваченных агентов ГОИ, хотя эта часть ИСД официально принадлежит Управлению безопасности в целях секретности. Отдел логистики
Департамент логистики отвечает за передачу природных, финансовых и человеческих ресурсов между объектами Фонда. Транспортировка осуществляется автомобильным, водным, железнодорожным, воздушным, космическим и другим транспортом. Имеется сложная система распределения приоритетов и классов важности перевозок; Объекты SCP и ресурсы, необходимые для их сдерживания, обычно получают высший приоритет.Устранение препятствий, возникающих при транспортировке, также является первоочередной задачей. Производственный отдел
Производственный отдел отвечает за выполнение любых материальных запросов, которые могут возникнуть во время поимки, содержания или любого другого процесса Фонда. Производственный отдел имеет несколько участков и зон, полностью посвященных созданию широкого профиля изделий на заказ или серийного производства. Проектирование и производство часто осуществляется на месте по соображениям безопасности.По тем же причинам ни производственный персонал, ни проектная документация никогда не покидают пределы объекта. Обычно они оснащены современным оборудованием и тщательно охраняются. Лечебное отделение
Медицинский отдел Фонда отвечает за поддержание хорошего здоровья сотрудников. Из-за характера работы Фонда медики часто сталкиваются с трудно поддающимися лечению и весьма необычными случаями ран, травм, инфекций и расстройств.В связи с этим ОД придерживается исключительно высоких кадровых стандартов. Некоторые медики Фонда специализируются на вторичных областях лечения, таких как меметические воздействия, психологические травмы и т. Д. Мобильные оперативные группы
Работа Фонда требует большого количества узкоспециализированных специалистов. Обычно это связано с аномалиями, которые требуют больших усилий и нестандартного подхода к локализации.Для решения этой проблемы создаются мобильные оперативные группы. MTF — это группы разного размера, которые специализируются на определенных ролях или реагируют на конкретные ситуации. У каждой группы есть название, состоящее из греческой буквы и числа; уточняющие префиксы и / или позывные могут использоваться в некоторых случаях. Дополнительную информацию можно найти в Обзоре мобильных оперативных групп. Научный отдел
Эффективная научная работа — краеугольный камень существования и деятельности Фонда.Научный отдел отвечает за изучение всех содержащихся и новых SCP-объектов, разработку мер противодействия событиям нарушения условий содержания и разработку различных устройств и лекарственных препаратов, которые будут производиться Производственным отделом. Сотрудники научного отдела набираются из числа лучших и перспективных ученых в различных областях. Отдел безопасности
Этот отдел отвечает за обеспечение безопасности и защиты всех помещений и объектов Фонда, независимо от их приоритета.Сотрудники SD обучены противодействию событиям нарушения условий содержания, вторжениям, саботажу, стихийным бедствиям и другим чрезвычайным ситуациям. Новички в этой услуге обычно проходят общее обучение перед тем, как попасть на объект или площадку. Специальное обучение работе с данным объектом проводится на выезде после задания. Департамент внутреннего трибунала
Отдел внутреннего трибунала Фонда SCP является институциональным судебным органом организации.Этот отдел, по просьбе ISD, рассматривает дела сотрудников Фонда и других лиц, которые подпадают под определение запрещенных Правовым кодексом Департамента внутреннего трибунала, включая такие записи, как: «I — Преступления против Фонда», «II — Преступления против нормальности и человечности »,« III — Существенные дисциплинарные нарушения »,« IV — Подготовка к проведению мероприятия класса K »,« V — Преступление государства и корпорации »,« VI — Прочие нарушения ». Трибунал может назначить различные приговоры по каждой категории нарушений (за исключением пятого, судебное разбирательство по которому ведется ГОК).Любое решение Трибунала может быть отменено Командованием O5. Вы можете ознакомиться с Положением о внутреннем трибунале Фонда SCP и его Юридическим кодексом здесь. Центральные базы данных мутаций и SNP
Репозиторий dbSNP-полиморфизмов NCBI, NIH, Bethesda Сервер молекулярной биологии ExPASy Швейцарский институт биоинформатики (SIB) Европейский институт биоинформатики EBI EMBL / EBI GeneSNPs (общедоступный Интернет-ресурс) Команда Центра генома Университета Юты ClinVar, NCBI Национальный центр биотехнологической информации Генатлас Жан Фрезаль, INSERM, Генетон, Франция, База данных генных карт М.Ребхан, Дж. Прилуски, В. Чалифа-Каспи и Дорон Ланцет Институт Вейцмана, Реховот, Израиль GVS: сервер изменения генома Д-р Дебора Никерсон, д-р Марк Ридер и команда США HGVbase: база данных вариаций генома человека Каролинский институт, Стокгольм, Швеция Европейский институт биоинформатики (EBI), Хинкстон, Великобритания и Европа, лаборатория молекулярной биологии (EMBL), Гейдельберг, Германия HOWDY: Организованная база данных полного генома человека Японская научно-техническая корпорация База данных мутаций генов человека, HGMD Д.Н. Купер, Э. В. Болл, П. Д. Стенсон М. Кравчак, Univ. Медицинского колледжа Уэльса. Кардифф, Великобритания jSNP: база База данных японских SNP Центр генома человека, Институт медицинских наук, Токийский университет База данных SNP Leelab Иризарри, К. Кустанович, В. Ли, К.Браун, Н. Нельсон, С. Вонг, В., Ли, К.Дж. UCLA Центр биоинформатики База данных человеческих SNP Институт Уайтхеда / Центр исследований генома Массачусетского технологического института Интернет-Менделирующее наследование в человеке, OMIM В. А. МакКусик и его коллеги, Университет Джона Хопкинса, NCBI NIEHS SNP ДокторДебора Никерсон, доктор Марк Ридер и команда США PharmGKB: База знаний по фармакогенетике и фармакогеномике Стэнфордский университет США SNP в Сиэтле Д-р Дебора Никерсон, д-р Марк Ридер и их команда Вашингтонского университета, Принстонского университета и Национального института сердца, легких и крови, U.
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-Y4738
мтДНК: T1a1
Расположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 660-780 CE
Y-ДНК: I-S7660
mtDNA: HV6
Местоположение: Hundstrup_Mose, Sealand, Дания
Возраст: Ранний викинг 670-830 CE
Y-ДНК: I-Y4051
mtDNA:
Образец: VK301 / Дания_Ladby Grav 4
Местоположение: Ladby, Funen, Дания
Возраст: Viking 640-890 CE
Y-ДНК: I-FT105192
mtDNA: R0a2b
Образец: VK306 / Sweden_Skara 33
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: I-FT115400
FTDNA Комментарий: Имеет 3 мутации с мужчина из Швеции.Образует новую ветвь I-S19291. Новая ветка = I-FT115400. VK151 не имеет покрытия для 2 из этих мутаций
мтДНК: h25a1
Образец: VK308 / Sweden_Skara 101
Расположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 века н.э.
Y-ДНК: R-BY33037
мтДНК: h2c
Образец: VK309 / Sweden_Skara 53
Местоположение: Варнем, Скара, Швеция
Возраст: Викинг 10-12 веков н.э.
Y-ДНК: R-YP6189
мтДНК: K1b1c
Образец: VK313 / Дания_Rantzausminde Grav 2
Местоположение: Ранцаусминде, Фюн, Дания
Возраст: Викинг 850-900 CE
Y-ДНК: R-JFS0009
мтДНК: h2b
Образец: VK315 / Дания_Bakkendrup Grav 16
Местоположение: Баккендруп, Силенд, Дания
Возраст: Viking 850-900 CE
Y-ДНК: I-Y98280
Комментарий FTDNA: Делится 1 SNP с мужчина из Нидерландов.Образует новую ветвь после I-Y37415 (P109). Новая ветвь = I-Y98280
мтДНК: T1a1b
Образец: VK316 / Дания_Hessum sk II
Местоположение: Хессум, Фюн, Дания
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y130659
Комментарий FTDNA: Сплит I- Y130594 (Z59). Получено для 1 предка для 6. Новый путь = I-Y130659> I-Y130594> I-Y130747. Древний образец STR_486 также принадлежит к этой группе, по I-Y130747
мтДНК: K1a4
Образец: VK317 / Дания_Kaargarden Grav BF99
Расположение: Kaagården, Langeland, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: J-BY62479
FTDNA Комментарий: 6-Splits J2 M67).Получено для 9, предков для 3. Новый путь = J-BY62479> J-BY72550
мтДНК: h3a2a1
Образец: VK320 / Дания_Bogovej Grav S
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-Y103013
Комментарий FTDNA: Делится 3 SNP с мужчина из Швеции. Образует новую ветвь вниз I-FT3562 (P109). Новая ветвь = I-Y103013
мтДНК: U5a1a1
Образец: VK323 / Дания_Рибе 2
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-S10185
мтДНК: K2a6
Образец: VK324 / Дания_Ribe 3
Расположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-BY16590
FTDNA Комментарий: Сплит R-BY16590 (L47).Получено для 7, предков для 3. Новый путь = R-S9742> R-BY16950
мтДНК: N1a1a1a2
Образец: VK326 / Дания_Рибе 5
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: R-Y52895
mtDNA: U5b1-T16189C! — T16192C!
Образец: VK327 / Дания_Рибе 6
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинг 9-11 века н.э.
Y-ДНК: I-BY463
мтДНК: H6a1a5
Образец: VK329 / Дания_Ribe 8
Местоположение: Рибе, Ютландия, Дания
Возраст: Викинги 9-11 веков н.э.
Y-ДНК: R-S18894
мтДНК: h4-T152C!
Образец: VK332 / Oland_1088
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-S8522
FTDNA Комментарий: Возможно, ниже I-BY195155.Имеет одну мутацию C> T с образцом BY195155 *
мтДНК: T2b24
Образец: VK333 / Oland_1028
Местоположение: Эланд, Швеция
Возраст: Viking 885 ± 69 CE
Y-ДНК: R-Z29034
mtDNA: h3a2a1
Образец: VK335 / Oland_1068
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н. Э.
Y-ДНК: R-BY39347
FTDNA Комментарий: Делится 8 SNP с мужчиной Франция.Образует новую ветвь вниз по R-BY39347 (U152). Новая ветвь = R-FT304388
мтДНК: K1b2a3
Образец: VK336 / Oland_1075
Местоположение: Эланд, Швеция
Возраст: Viking 853 ± 67 CE
Y-ДНК: R-BY106906
мтДНК: K2a3a
Образец: VK337 / Oland_1064
Местоположение: Эланд, Швеция
Возраст: Viking 858 ± 68 CE
Y-ДНК: I-BY31739
FTDNA Комментарий: Возможный Z140
mtDNA1b: 9003a U5a
Образец: VK338 / Дания_Bogovej Grav BV
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-A6707
мтДНК: W3a1
Образец: VK342 / Oland_1016
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-BY78615
FTDNA Комментарий: Имеет 2 SNP с мужчиной из Финляндия.Образует новую ветвь вниз I2-Y23710 (L801). Новая ветвь = I-BY78615
мтДНК: h3a1
Образец: VK343 / Oland_1021
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Y7232
мтДНК: h4h
Образец: VK344 / Oland_1030
Расположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-BY32357
mtDNA: J1c2t
Образец: VK345 / Oland_1045
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: R-FT148754
FTDNA Комментарий: Сплит R-FT148754 (DF63) .Получено для 8, предков для 6. Новый путь = R-FT148796> R-FT148754
мтДНК: h5a1
Образец: VK346 / Oland_1057
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 века н.э.
Y-ДНК: J-Z8424
мтДНК: h3a2b
Образец: VK348 / Oland_1067
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-Z171
мтДНК: T2b28
Образец: VK349 / Oland_1073
Местоположение: Оланд, Швеция
Возраст: Viking 829 ± 57 CE
Y-ДНК: R-BY166065
FTDNA Комментарий: Делится 2 SNP с мужчиной из Англии .Образует ответвление вниз от R-BY166065 (L1066). Новая ветвь = R-BY167052
мтДНК: h2e2a
Образец: VK352 / Oland_1012
Местоположение: Эланд, Швеция
Возраст: Викинг 9-11 веков н.э.
Y-ДНК: I-FGC35755
FTDNA Комментарий: Возможно, образует ветвь вниз I -Y15295. 2 возможные мутации G> A с образцом I-Y15295 *
мтДНК: H64
Образец: VK354 / Oland_1026
Местоположение: Эланд, Швеция
Возраст: Viking 986 ± 38 CE
Y-ДНК: R-S6752
мтДНК: h3a1
Образец: VK355 / Oland_1046
Местоположение: Эланд, Швеция
Возраст: Викинг 847 ± 65 CE
Y-ДНК: L-L595
FTDNA Комментарий: Присоединяется к 2 другим древним на этой редкой ветви .ASH087 и I2923
мтДНК: U5b1b1a
Образец: VK357 / Oland_1097
Местоположение: Эланд, Швеция
Возраст: Viking 1053 ± 60 CE
Y-ДНК: I-FT49567
FTDNA Комментарий: Делится 4 SNP с мужчиной из Англии . Образует новую ветвь I-A5952 (Z140). Новая ветвь = I-FT49567
мтДНК: J2b1a
Образец: VK362 / Дания_Bogovej LMR 12077
Расположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: E-CTS5856
FTDNA Комментарий:
3 Возможно 90 E-Z мтДНК: V7b
Образец: VK363 / Дания_Bogovej BT
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY198083
Комментарий FTDNA: Делится 2 SNP с мужчиной из Швейцарии.Образует новую ветвь I-A1472 (Z140). Новая ветвь = I-BY198083
мтДНК: U4b1a1a1
Образец: VK365 / Дания_Bogovej BS
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: R-BY34800
мтДНК: U8a2
Образец: VK367 / Дания_Bogovej D
Местоположение: Богёвей, Лангеланд, Дания
Возраст: Викинг 10 век н.э.
Y-ДНК: I-BY67827
Комментарий FTDNA: VK506 и VK19967 9 Внутренние отделы — SCP Foundation




 01.83;
01.83; Учитываются также виды заполнителя смеси.
Учитываются также виды заполнителя смеси.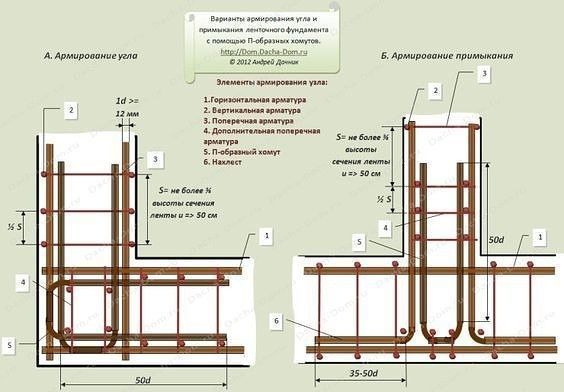
 13330.2012 «Каменные и армокаменные конструкции. Актуализированная редакция СНиП II-22-81»
13330.2012 «Каменные и армокаменные конструкции. Актуализированная редакция СНиП II-22-81» biotech.cornell.edu/brc/genomic-diversity
biotech.cornell.edu/brc/genomic-diversity На ПК это CTRL + F, чтобы отобразить поле поиска. Если ваша гаплогруппа не отображается, возможно, вы ниже по течению от гаплогруппы Viking, поэтому вам нужно будет использовать дерево блоков ДНК Y (для тестеров большого Y) или публичное гаплотерево здесь.
На ПК это CTRL + F, чтобы отобразить поле поиска. Если ваша гаплогруппа не отображается, возможно, вы ниже по течению от гаплогруппы Viking, поэтому вам нужно будет использовать дерево блоков ДНК Y (для тестеров большого Y) или публичное гаплотерево здесь. Если вы не заказывали тест митохондриальной ДНК, вы можете заказать или обновить его здесь.
Если вы не заказывали тест митохондриальной ДНК, вы можете заказать или обновить его здесь.